
代谢组学技术服务-非靶向代谢组学

非靶向代谢组学旨在发现具有差异代谢物及其所处代谢径。它以组学的视角无偏向性地检测所有的内源性小分子代物,并在代谢层面发现具有差异的代谢物(潜在的生物标物)及其所处代谢途径,进而分析和探索引起这种生命现象或病理现象背后的代谢机制。并采用生物信息学手段研究生物受到扰动(如基因改变或环境变化)后内源性代谢物的整体谢特征或变化规律。与传统的数个代谢物测定或靶向代谢组学定量相比,全谱代谢组学的特点在于对所有的内源性小分谢物(通常是浓度较高的代谢物)同时进行测定,因此得到的代谢信息更客观全面,从而避免研究的方向性错误。
我们的优势:
- 无便向性,适用于各类型样本和代谢物;
- 适合于复杂基质,单次检测可获得更多代谢物和代谢网络信息;
- 自建3500+常见物质标品数据库+购买各种商业数据库+公共数据库,超过十万种代谢物信息;
- 全程人工校对,机器和人工只能辅助,使用保留时间、保留指数、质核比和多级碎片离子信息进行物质定性,结果更准,物质数量更多。
| 样本名称 | 最低量 | 一般量 |
| 动物和临床各类组织样本 | 5-10mg | 50mg |
| 血样(血清、血浆和全血) | 10μl | 50μl |
| 尿样 | 10μl | 50μl |
| 粪便和肠道内容物 | 10mg | 25mg |
| 体液样本(脑脊液和唾液等) | 5-10μl | 25μl |
| 植物组织样本(根、茎、叶、花和果实等) | 10mg | 50mg |
| 细胞和微生物菌体 | 1*10^5cells | 1*10^6cells |
| 培养基和发酵液 | 10μl | 50μl |
1.数据检查
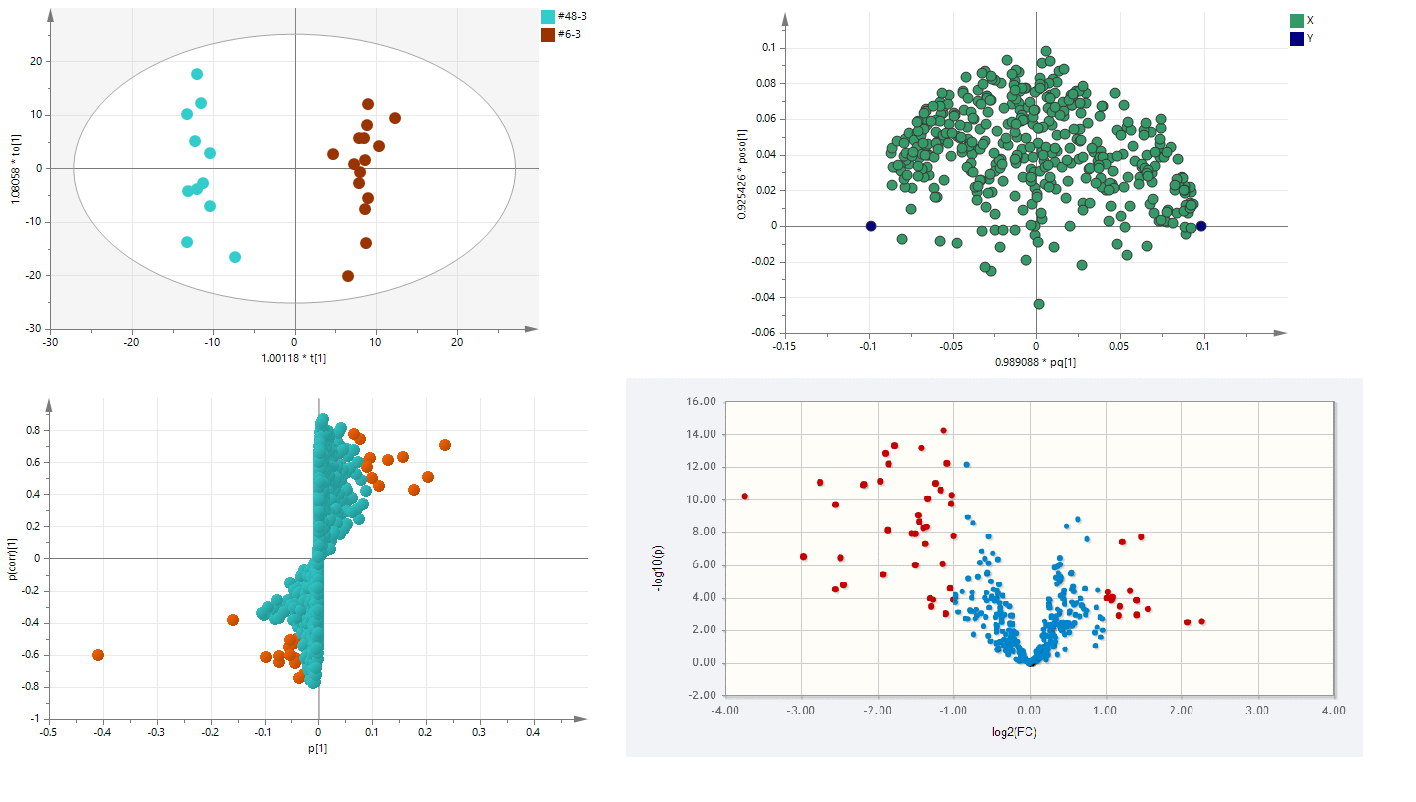
对所有样本的总离子流色谱图(TIC)色谱图进行可视化检查,如图1:

2.数据预处理:
讲仪器检测得到的原始数据转化为通用格式,然后对质谱数据进行峰匹配,峰对齐和保留时间校正,得到去卷积的质谱数据。然后,对来自于同一个物质的各个峰数据进行归属分析。
3.多维统计分析:
对数据进行后处理,将处理后的数据导入到SIMCA软件进行多维统计分析,在软件中首先进行Pareto格式化(par scaling)和平均中心化(mean-centering)处理,然后再进行PCA、PLS-DA和OPLS-DA等多维统计分析。
● 3.1整体PCA分析和组件PLS-DA
采用SIMCA软件对整体样本进行PAC分析(主成分分析),用于解释和分析各组样本之间的代谢差异。如图2:整体PCA分析

对样本进行PLS-DA分析(偏最小二乘法判别分析),用于分析组间的差异代谢物信息的显著性,并进行Permutation test (置换检验),结合模式的Q2和R2Y,验证模型的可靠性。如图3:组间样本PLS-DA分析和Permutation test

● 3.2 组件OPLS-DA分析和volcano plot分析
为消除无关噪音信息及准确获得两组样本间的显著性差异代谢物信息,我们采用组间OPLS-DA(正交偏最小二乘法判别分析)进行分析,进而获得OPLS-DA模型和相关值(VIP值),作为下一步进行差异物定性筛选的参考。并可对OPLS-DA分析的结果进行Loading Plot 分析和S-plot 分析,另外使用R语言平台进行Volcano Plot (火山图)分析。如图4:组件样本OPLS-DA分析和volcano plot分析、S-plot分析和Volcano Plot分析
 4.单维统计分析
4.单维统计分析
对数据进行单维统计分析,dui对数据分别jin'xing进行Shapiro Wilk's test、Welch's Test和Wilcoxon Mann-Whitney test(U test)。呈正态分布的变量则采用Wilk's test结果,而呈非正态分布的变量则采用Wilcoxon Mann-Whitney test结果,综合得到各变量在各对比组之间的显著性分析结果(p-value)
5.差异代谢物定性
多维统计分析(VIP>1)结合单维统计(p-value<0.05)寻找差异性表达代谢物,
采用软件人工结合的方式,使用RT和特征M/Z(GC-MS)或精确分子量和二级质谱(LC-MS)与数据库进行逐一对比分析,差异性代谢物的定性方法为:搜索自建的标准物质数据库、Fiehn GC/MS Metabolomics RTL Library 、Golm Metabolome Database、Metlin、HMDB、KEGG、Lipid和NIST等商业数据库。差异物列表示例如表1:组间样本的差异性代谢物示例
| Metabolites | p-value | VIP | FC(A/B) | HMDB | KEGG | Pathway(KEGG) |
| pyruvic acid | 2.16E-03 | 1.71 | -0.51 | HMDB00243 | C00022 | Glycolysis/Gluconegenesis;Citrate cycle(TCA cycle);Pentose phosphate pathway |
| glucose | 4.11E-02 | 1.82 | 2.65 | HMDB00122 | C00031 | Glycolysis/Gluconegenesis;Pentose phosphate pathway;Galactose metabolism |
| gluconic acid | 1.52E-02 | 1.63 | 0.68 | HMDB00625 | C00257 | Pentose phosphate pathway |
| mannitol | 4.11E-02 | 1.45 | 1.47 | HMDB00765 | C00392 | Fructose and mannose metabolsm |
| dulcitol | 2.16E-03 | 1.74 | 3.72 | HMDB00107 | C01697 | Galactose metabolism |
| galactonic acid | 2.16E-03 | 1.96 | 2.24 | HMDB00565 | C00880 | Galactose metabolism |
| ethanolamine | 2.16E-03 | 2.10 | 1.00 | HMDB00149 | C00189 | Glycerophospholopid metabolism |
6.相关性分析
●6.1 Pearson Correlation 分析
为了表征各差异性代谢物之间的(浓度)相关性,我们会对这些物质的定量信息经行Pearson Correlation分析。如图5:差异性代谢物的相关性矩阵图

●6.2热图分析:
为了表示差异物之间的聚类关系,我们会对这些物质的定量信息进行热图分析,如图6:差异性代谢物的热图

7.代谢通路分析
●7.1代谢通路进行归类分析
我们采用KEGG数据库对每个差异代谢物所属的代谢通路进行归类分析,如图7:差异性代谢物所属KEGG代谢通路实例

●7.2metametabonanalyst pathway分析
我们用软件metametabonanalyst对差异性代谢物进行pathway analysis,metabolome view如图8所示:

Pathway views如表2所示,total表示该途径所含的代谢物总数,hits表示该途径含有差异性代谢物数,-log(p)表示图8纵坐标值,impact表示图8横坐标值。参数如表2组间样本的pathway view所示:
| Pathyway | Total | Expected | Hits | Raw p | -log(p) | Holm adjust | FDR | Impact |
| Pantothenate and CoA biosynthesis | 16 | 0.39402 | 2 | 0.05672 | 2.8695 | 1 | 1 | 0 |
| Zeatin biosynthesis | 16 | 0.39402 | 2 | 0.05672 | 2.8695 | 1 | 1 | 0 |
| Butanoate metabolism | 20 | 0.49252 | 2 | 0.08457 | 2.4702 | 1 | 1 | 0 |
| Alanine,aspartate and glutamate metabolism | 21 | 0.51715 | 2 | 0.09208 | 2.3851 | 1 | 1 | 0 |
| C5-Branched dibasic acid metabolism | 4 | 0.098505 | 1 | 0.09504 | 2.3534 | 1 | 1 | 0 |
| Citrate cycle(TCA cycle) | 20 | 0.49252 | 1 | 0.39526 | 0.9282 | 1 | 1 | 0 |
| Pyruvate metabolism | 20 | 0.49252 | 1 | 0.39526 | 0.9282 | 1 | 1 | 0.1148 |
| Purine metabolism | 55 | 1.3544 | 2 | 0.39526 | 0.9244 | 1 | 1 | 0.065 |
定制化分析
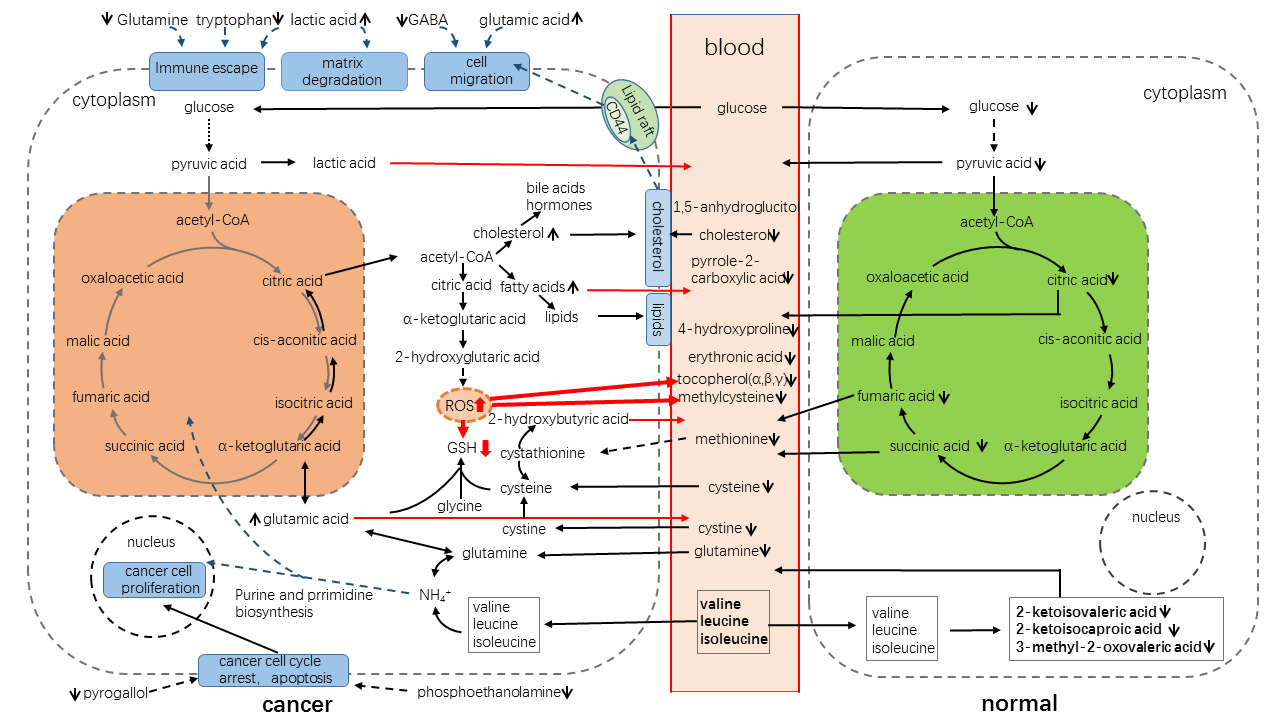
我们根据客户的实验结果,集合各方面现有成果,进行定制化的代谢通路分析,如图9定制化代谢通路分析图示例:
2.其他定制分析
如s使用Cytoscape软件对样本的代谢组、蛋白组、基因组进行关联分析(如图10),包括但不限于多组学关联分析、多平台数据整合分析等根据客户实际要求,我们指定详细的方案进行科学合理的分析。只要是基于代谢组学技术的所有统计分析,我们都可以提供高质量技术服务。


主要服务产品分为非靶向代谢组学(传统优势服务项目)、精准的靶向定量代谢组学(业内最全、特色服务项目)、精细化的脂质组学、基于稳定性同位素标记技术的代谢流组学(业内仅有、特色)和领先数据分析服务。自建的包含3000+代谢物信息的气相色谱质谱和液相色谱质谱代谢物标准品数据库,配合Golm代谢组数据库、Fiehn代谢组数据库、NIST数据库和脂质理论库等商业和公共数据库,可提供代谢物准确定性定量服务。其中,谱领靶向定量代谢组学技术服务项目种类最多,形成了品牌,在国内居于领军位置。
2016年,谱领代谢组学技术服务在行业内率先通过ISO9001:2008质量管理体系认证;2017年,谱领生物商标正式获得国家商标局注册证书,开启了品牌战略步伐。本着“专注、创新、精准、领先”的科学理念,谱领生物持续创新,致力于为生物医药领域科学研究和产品开发提供专家级代谢组学整体解决方案。谱领生物在临床大样本代谢组学研究,尤其在实验测试标准化、质量控制和数据校正等方面拥有丰富的经验。谱领代谢组学平台已助力客户发表了多篇高影响因子论文,数篇论文影响影子达到30分左右。迄今已为欧美和国内两百多所高校、科研院所、医院和企业提供专家级的代谢组学技术服务,获得国内外客户广泛高度评价。
企业文化:企业理念:秉承以学术研究为核心,以服务体验为中心,以达到促进学术研究与学术创新的最终目的为己任的义务。
价值观:追求卓越,鼓励探索。
愿景:提供一流的一站式转化组学方法学研究、方法开发综合解决方案。
使命:通过高性价比、高效率的创新型服务和一流售后追踪服务帮助广大客户解决学术研究上的系统性问题,缩短整个项目周期、降低实验成本。


| 单位名称: |
|
详细地址:
上海市嘉定区宝安公路4997号国际医疗产业园1号楼
|
|
qq:
|
| 官网: |
| 联系电话: |
| Email: |




