深度神经网络助力对肿瘤细胞鉴定及空间恶性区域的识别
2024-07-29 来源:本站 点击次数:1704在肿瘤相关单细胞转录组测序中,肿瘤细胞的甄别一直是一个让人头疼的问题。CNV可以用于区分肿瘤细胞和正常细胞,常用的inferCNV方法往往需要一个清晰的非肿瘤细胞注释做为参考去佐证肿瘤细胞,CopyKAT对于细胞数量多的分析项目是极不友好的,时间成本消耗也是极高的。下面介绍一种新的肿瘤细胞预测的方法:Cancer-Finder,其可以应用于肿瘤细胞的鉴定和空间恶性区域的识别。
Cancer-Finder是一种基于深度学习算法,用于单细胞转录组数据肿瘤细胞预测和空间转录组数据恶性区域的识别的软件,其平均预测精准度可以高达95.16%。Cancer-Finder收集13种不同肿瘤相关组织的单细胞数据集做为训练数据集对肿瘤细胞进行预测。
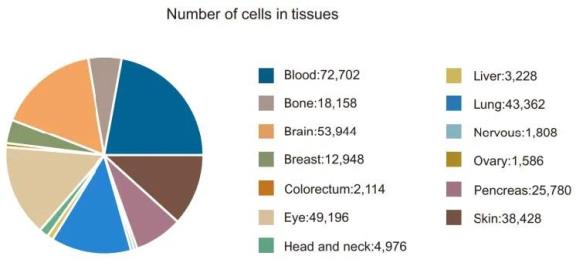
与当前一些肿瘤细胞预测手段,诸如:CaSee、CopyKAT、SCEVAN、ikarus等比较,Cancer-Finder分析时间花费的更少,效果更佳。Cancer-Finder使用Python语言编写,也有着对计算机资源消耗相对较少的优点。
如何安装?
基于Linux的软件安装:
1. 首先,下载Cancer-Finder到本地,并解压,进入该目录,你可看到如下文件结构,infer.py为预测肿瘤细胞程序。
2. 然后,使用Conda安装 Cancer-Finder。
a) 首先创建一个名字叫csf的虚拟环境并安装3.9.16版本的python软件:
conda create -n scf python==3.9.16
b) 然后激活scf虚拟环境:conda activate scf
c) 最后使用pip安装Cancer-Finder所需模块:
pip install -r requirements.txt -i
pip install matplotlib==3.8 -i
d) 注意事项:这里推荐使用Python的清华镜像(-i 参数),安装速度较快,matplotlib建议使用3.8版本。以上安装步骤亲测可用。
如何运行预测程序?
执行下面一行代码即可开启分析。
python -u infer.py --ckp=../sc_pretrain_article.pkl --matrix=sample_data/sample_data_matrix.tsv --out=out.csv
--ckp参数:用于预测肿瘤细胞或者区域的预训练好的模型数据,存储为.pkl格式文件。Cancer-Finder提供了预训练完成的单细胞模型数据 sc_pretrain_article.pkl 和空间转录组模型数据 st_pretrain_article.pkl。
--matrix参数:待注释的表达矩阵文件,支持tsv、csv、h5ad格式的文件。
--out 参数:结果输出。
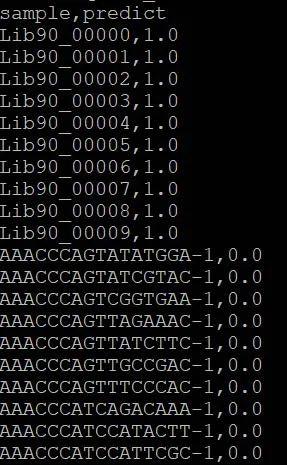
输出结果一共两列,如下图,第一列为细胞信息,第二列为细胞对应预测结果信息,1.0代表被预测为肿瘤细胞,0.0代表被预测为非肿瘤细胞。从结果上看这里的目标是只需区分肿瘤细胞和非肿瘤细胞即可,其本质是机器学习中的深度神经网络解决二元分类问题。
如何训练自己的模型数据?
Cancer-Finder提供的预训练模型数据(单细胞和空间转录组模型数据)可能并不适用于某些领域的研究,所以提供了训练私有模型的程序 train.py 。
python -u train.py \
--train_dir=\
--val_dir=\
--batch_size=\
--lr=\
--max_epoch=\
--output=\
--gpu_id=
数据结构详细请参考下载到本地的Cancer-Finder软件包 data目录。
实际效果如何?
使用GEO数据(GSE155446)比较预测结果。
python -u infer.py --ckp=sc_pretrain_article.pkl --matrix=GSE155446_human_raw_counts.csv --out=GSE155446_human.rs.tsv
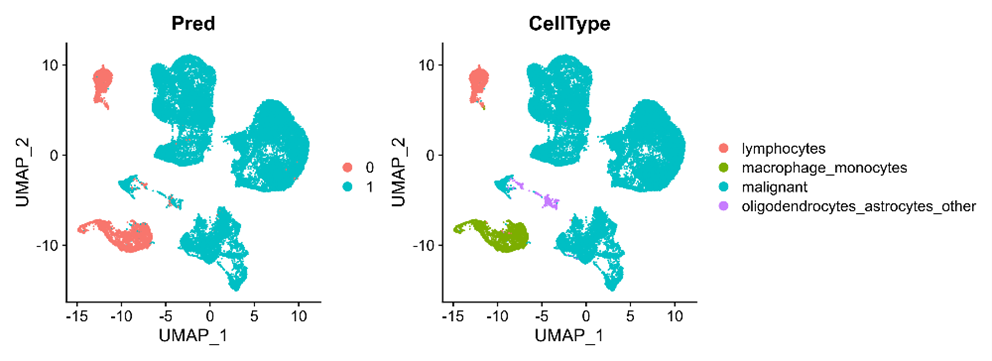
上图中Pred标签UMAP图为使用Cancer-Finder对肿瘤细胞的预测结果的展示,1代表肿瘤细胞,0代表非肿瘤细胞;CellType标签的UMAP图为数据集提供的注释结果展示。从UMAP图上可以清晰看出Cancer-Finder预测的肿瘤细胞与注释高度一致。接下来看下注释结果与预测结果的比例情况,如下图。
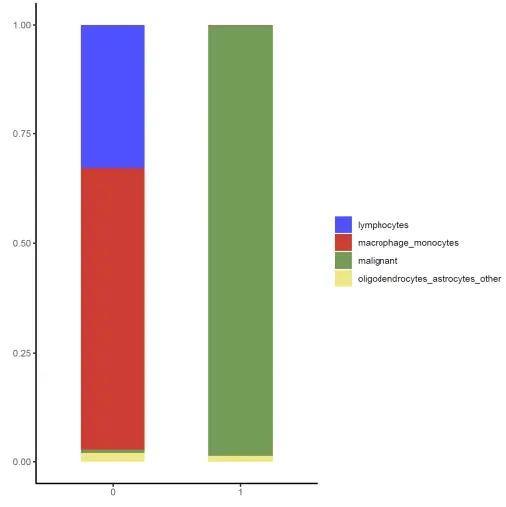
从上图可以看出Cancer-Finder识别出了极高比例的肿瘤细胞,从这个案例可以看出Cancer-Finder的效果是很好的,此数据接近4w的细胞数量,在几分钟内可以完成肿瘤细胞的识别,有着相当不俗的表现。在已知背景数据集中Cancer-Finder的预测能力是极好的,对于未知领域的数据可以综合不同的算法交叉比较,大概率应该对个性化研究也会有着不错的结果。
对于空间转录的数据,大家可以小试牛刀。
参考文献:
Zhong, Z., Hou, J., Yao, Z. et al. Domain generalization enables general cancer cell annotation in single-cell and spatial transcriptomics. Nat Commun 15, 1929 (2024).

