小鼠单细胞图谱开源数据库Tabula Muris在单细胞测序的应用
2021-06-17 来源:腾泉生物 点击次数:6401
来自斯坦福大学、陈-扎克伯格生物中心以及加州大学旧金山分校的研究人员建立了一个名为Tabula Muris的开源数据库,收录了小鼠的多个细胞类型及其基因表达数据。这项成果发表在《Nature》杂志上。研究团队利用流式分选(FACS)方法和微流体液滴捕获(droplet)方法,从小鼠20个不同器官中分离出超过10万个细胞,并开展单细胞RNA测序。这些数据让人们能够比较各个细胞类型和组织间的基因表达,从而更深入地了解细胞多样性。

实验方法
小鼠组织和器官来源:
主动脉、膀胱、骨髓、大脑、膈肌、脂肪、心脏、肾脏、大肠、四肢肌肉、肝脏、肺、乳腺、胰腺、皮肤、脾脏、胸腺、舌头和气管等。
数据来源于两种不同单细胞测序方法:
• 10X: 细胞个数n=55656 基于油包水微流控(droplet)技术的3’端测序:能够在相对低的覆盖率下对相应器官来源的数千个细胞完成高细胞通量的检测。
• Smart-seq2: 细胞个数n=44949 基于流式分选(FACS) plate-based的RNA全长测序:提供灵敏度和覆盖度更高数据。
该研究中基于FACS的SMART-seq2单细胞测序流程中,cDNA合成扩增、文库制备和文库pooling分别使用了SPT Labtech的dragonfly discovery、mosquito LV genomics以及mosquito X1进行移液操作。

实验结果
对不同器官组织来源的细胞进行分类,利用t-SNE算法来观察各器官的不同细胞类型之间的关系。
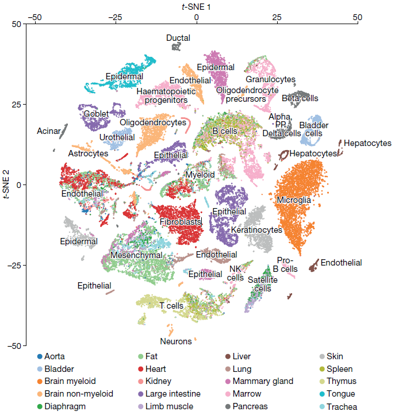
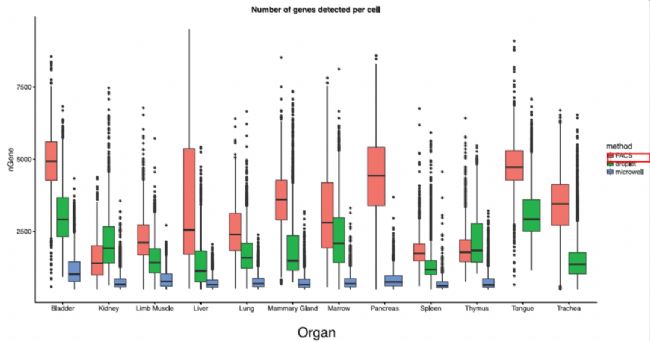
对不同的单细胞测序方法进行比较,结果发现在不同的器官类型中FACS法检测到的基因数量最多,几乎是droplet的两倍。
总结
在单细胞研究中, 利用高通量的10X与高灵敏度的Smart-seq2相结合,对于不同的研究方向具有互补性的优势:
• 基于droplet技术的优势是测的细胞数量多能够发现稀少的细胞和细胞状态。
• 基于FACS方法的特点是测的细胞数量少但是覆盖度高、能够富集特定的细胞,可用于研究微异质性(包括低表达基因,可变剪接,序列变异分析)。
对于基于FACS的Smart-seq2单细胞测序,往往具有一些难点:
1. 单细胞建库试剂的成本昂贵
2. 需要对每个细胞单独建库,操作繁琐,难以上量
而来自SPT Labtech最新的液体处理技术,可以将基于FACS的单细胞测序,往更低成本、更高通量、更自动化的方向建设工作流程。

参考文献:
Consortium, T. M. , et al. "Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris." Nature 2018.

相关文章
更多 >

