探秘表观遗传的技术原理及适用场景介绍
2025-05-19 来源:本站 点击次数:587经典遗传学理论认为,DNA碱基序列作为遗传信息的载体可以将亲代性状遗传给子代。近年来,随着对染色质和组蛋白的深入研究,表观遗传学逐渐兴起。表观遗传是指在DNA序列不发生改变的前提下,基因表达和表型发生可遗传变化的现象。在表观遗传研究领域,选择合适的研究技术至关重要。DAP-seq、ChIP-seq、CUT&Tag、ATAC-seq是表观遗传研究的常用技术,该如何选择呢?接下来,小编将深入剖析这些技术,助您找到表观遗传研究的得力“助手”。
一、技术原理
☞ DAP-seq:体外探索蛋白-DNA互作的先锋
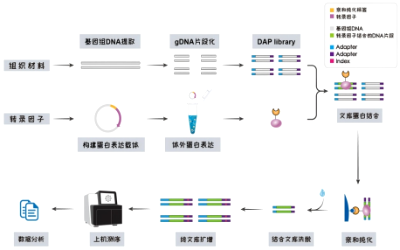
DAP-seq,全称为DNA亲和纯化测序(DNA Affinity Purification Sequencing),是一种基于体外重组蛋白的转录因子结合位点鉴定技术。该方法通过将体外表达的转录因子和基因组DNA进行亲和纯化,然后洗脱与转录因子结合的DNA片段,进行高通量测序,生成转录因子的全基因组结合位点图谱。DAP-seq打破了传统ChIP-seq对抗体的依赖性,适用于非模式生物及抗体难以获取的转录因子研究。同时由于采用体外结合体系,可保留DNA甲基化等表观遗传修饰对蛋白-DNA互作的影响,为表观遗传研究提供技术支持。
一、技术原理
☞ DAP-seq:体外探索蛋白-DNA互作的先锋
DAP-seq,全称为DNA亲和纯化测序(DNA Affinity Purification Sequencing),是一种基于体外重组蛋白的转录因子结合位点鉴定技术。该方法通过将体外表达的转录因子和基因组DNA进行亲和纯化,然后洗脱与转录因子结合的DNA片段,进行高通量测序,生成转录因子的全基因组结合位点图谱。DAP-seq打破了传统ChIP-seq对抗体的依赖性,适用于非模式生物及抗体难以获取的转录因子研究。同时由于采用体外结合体系,可保留DNA甲基化等表观遗传修饰对蛋白-DNA互作的影响,为表观遗传研究提供技术支持。
☞ ChIP-seq:体内捕捉蛋白-DNA结合的猎手
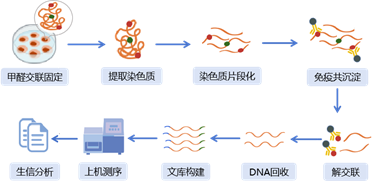
ChIP–seq,全称是染色质免疫共沉淀测序(Chromatin Immunoprecipitation followed by Sequencing),是在体内分离与特定蛋白结合的DNA片段的经典方法。通过甲醛固定细胞内DNA-蛋白质复合物,再经超声或酶解将染色质破碎成小片段;随后,利用目标蛋白(如转录因子、修饰组蛋白等)的特异性抗体,免疫沉淀蛋白DNA复合物,经洗涤、解交联后回收DNA;构建文库并高通量测序,将序列与参考基因组比对,即可精准定位目标蛋白结合位点。ChIP-seq能够真实反映细胞内蛋白质与DNA的相互作用情况,为揭示基因表达调控机制提供关键线索。
ChIP–seq,全称是染色质免疫共沉淀测序(Chromatin Immunoprecipitation followed by Sequencing),是在体内分离与特定蛋白结合的DNA片段的经典方法。通过甲醛固定细胞内DNA-蛋白质复合物,再经超声或酶解将染色质破碎成小片段;随后,利用目标蛋白(如转录因子、修饰组蛋白等)的特异性抗体,免疫沉淀蛋白DNA复合物,经洗涤、解交联后回收DNA;构建文库并高通量测序,将序列与参考基因组比对,即可精准定位目标蛋白结合位点。ChIP-seq能够真实反映细胞内蛋白质与DNA的相互作用情况,为揭示基因表达调控机制提供关键线索。
☞ CUT&Tag:高效灵敏的蛋白-DNA互作探测仪
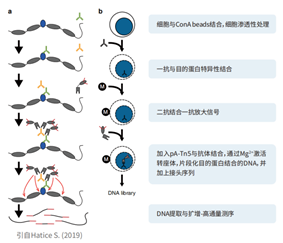
CUT&Tag,即靶向剪切及转座酶技术(Cleavage Under Targets and Tagmentation),是用来研究组蛋白修饰以及蛋白质与DNA之间的相互作用的新锐技术。在进行CUT&Tag实验时,刀豆素A磁珠(ConA磁珠)通过特异性识别细胞表面糖基化分子,将细胞牢牢固定。依次加入目标蛋白特异性抗体与二抗,结合目的蛋白并放大信号。随后引入Protein A/G-Tn5融合转座酶,这个融合酶就像是一个自带“剪刀”和“标签”的分子机器。Protein A/G能够与抗体特异性结合,引导Tn5转座酶精准地定位到目标蛋白结合的染色质区域,而Tn5转座酶则会在切割DNA的同时,将测序接头直接插入到切割位点两端,完成对DNA片段的标记。最后,仅需简单PCR扩增,即可快速构建高通量测序文库。
相较传统ChIP-seq技术,CUT&Tag实验周期从3-5天缩短至1-2天,细胞用量从百万级降至千级,同时显著降低背景噪音,尤其适用于微量样本的高灵敏度蛋白-DNA互作研究,为生命科学领域打开了新的研究窗口。
相较传统ChIP-seq技术,CUT&Tag实验周期从3-5天缩短至1-2天,细胞用量从百万级降至千级,同时显著降低背景噪音,尤其适用于微量样本的高灵敏度蛋白-DNA互作研究,为生命科学领域打开了新的研究窗口。
☞ ATAC-seq:打开染色质可及性大门的钥匙
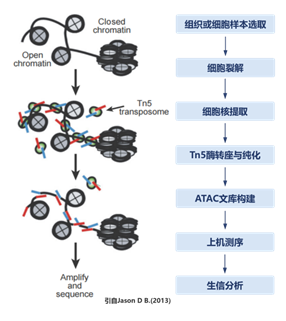
ATAC-seq,染色质可及性测序(Assay for Transposase-Accessible Chromatin using sequencing),是探索全基因组染色质可及性的前沿技术。其核心原理基于转座酶对开放染色质区域的偏好性。在细胞内,DNA 与组蛋白缠绕形成染色质,而染色质的松紧状态直接决定了DNA片段能否被转录因子等调控蛋白识别。那些处于松散、开放状态的染色质区域,正是基因表达调控的“活跃地带”。
ATAC-seq借助高活性的Tn5转座酶突变体,精准定位基因组中的开放染色质区域。携带特定 DNA 序列标签的Tn5转座复合物与细胞核共同孵育时,转座酶会切割开放染色质区域的DNA,并快速为切割位点两端添加测序接头,随后进行PCR扩增和高通量测序,最后通过分析测序数据中reads的分布,即可清晰描绘全基因组染色质的开放图谱,锁定潜在的基因调控位点与转录因子结合区域。该技术具有操作简便、耗时短(3小时内即可完成实验准备)、样本需求量少等优势,同时可支持单细胞或微量样本分析。
ATAC-seq借助高活性的Tn5转座酶突变体,精准定位基因组中的开放染色质区域。携带特定 DNA 序列标签的Tn5转座复合物与细胞核共同孵育时,转座酶会切割开放染色质区域的DNA,并快速为切割位点两端添加测序接头,随后进行PCR扩增和高通量测序,最后通过分析测序数据中reads的分布,即可清晰描绘全基因组染色质的开放图谱,锁定潜在的基因调控位点与转录因子结合区域。该技术具有操作简便、耗时短(3小时内即可完成实验准备)、样本需求量少等优势,同时可支持单细胞或微量样本分析。
二、技术特点
为了更直观地展现这四种技术的差异,我们通过以下表格来详细对比它们在样本需求、灵敏度、实验周期、成本等方面的特点:
为了更直观地展现这四种技术的差异,我们通过以下表格来详细对比它们在样本需求、灵敏度、实验周期、成本等方面的特点:
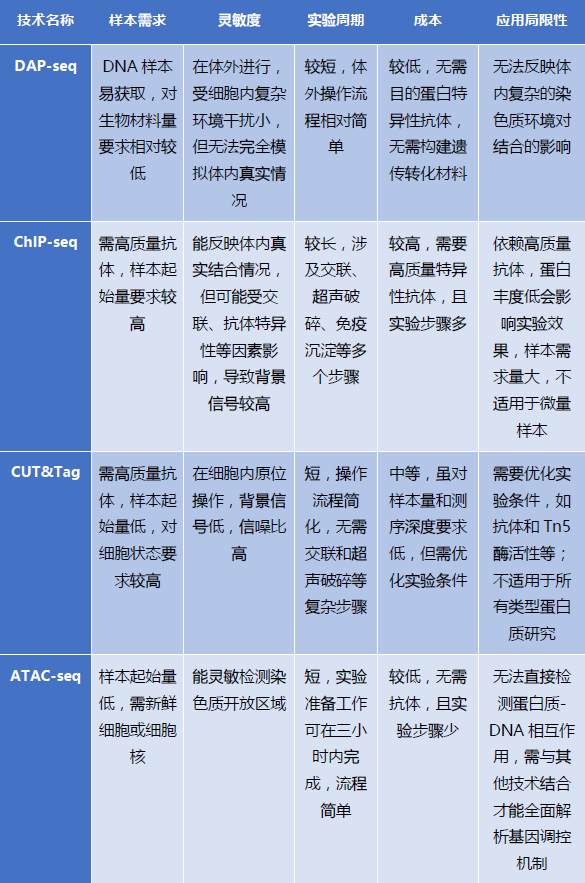
三、适用场景
在实际的表观遗传研究中,选择合适的技术就如同为不同的旅程挑选合适的交通工具,需要根据具体的研究目标、样本类型和资源条件等因素来综合考量。
☞ DAP-seq:非模式生物与转录因子研究的得力助手
DAP-seq在解析转录因子与DNA结合位点上独具优势,特别适用于难以获取高质量抗体的样本,尤其是非模式生物研究。在植物领域,许多物种缺乏成熟抗体资源,DAP-seq技术为此开辟了新路径
☞ ChIP-seq:全面解析蛋白质-DNA相互作用的经典之选
染色质免疫共沉淀测序(ChIP-seq)是研究蛋白质与DNA相互作用的经典技术,适用于样本充足且具备高质量特异性抗体的实验场景。该技术能够在体内生理环境下,系统解析蛋白质与DNA的结合图谱,为基因调控网络研究提供关键信息。
☞ CUT&Tag:微量样本与单细胞研究的利器
CUT&Tag技术以低样本量需求和高灵敏度的显著优势,成为微量样本及单细胞研究的核心工具。该技术能在单细胞层面高分辨率解析蛋白质与DNA的互作关系,为探究细胞异质性及发育进程中的基因调控奥秘提供了关键手段。
☞ ATAC-seq:染色质开放性与基因调控元件研究的先锋
ATAC-seq聚焦染色质开放性研究,可高效探索全基因组染色质开放区域与潜在调控元件,凭借样本需求量少、检测灵敏快速的优势,成为解析基因表达调控的重要技术。
☞ 技术组合:发挥协同优势
通常单一技术难以满足复杂研究需求,技术组合可提供更全面的研究视角。ATAC-seq与ChIP-seq、CUT&Tag或DAP-seq结合,能同时获取染色质开放性与蛋白质-DNA结合位点信息,帮助研究者深入解析基因调控网络。ATAC-seq与RNA-seq联合使用,可关联染色质开放性与基因表达变化,助力探索基因表达调控机制。
四、总结
DAP-seq、ChIP-seq、CUT&Tag、ATAC-seq四种技术在表观遗传研究中各展所长,其原理、特点与适用场景各异,我们需要结合具体研究需求和条件,才能做出合适的选择。欢迎感兴趣的同学前来咨询!
在实际的表观遗传研究中,选择合适的技术就如同为不同的旅程挑选合适的交通工具,需要根据具体的研究目标、样本类型和资源条件等因素来综合考量。
☞ DAP-seq:非模式生物与转录因子研究的得力助手
DAP-seq在解析转录因子与DNA结合位点上独具优势,特别适用于难以获取高质量抗体的样本,尤其是非模式生物研究。在植物领域,许多物种缺乏成熟抗体资源,DAP-seq技术为此开辟了新路径
☞ ChIP-seq:全面解析蛋白质-DNA相互作用的经典之选
染色质免疫共沉淀测序(ChIP-seq)是研究蛋白质与DNA相互作用的经典技术,适用于样本充足且具备高质量特异性抗体的实验场景。该技术能够在体内生理环境下,系统解析蛋白质与DNA的结合图谱,为基因调控网络研究提供关键信息。
☞ CUT&Tag:微量样本与单细胞研究的利器
CUT&Tag技术以低样本量需求和高灵敏度的显著优势,成为微量样本及单细胞研究的核心工具。该技术能在单细胞层面高分辨率解析蛋白质与DNA的互作关系,为探究细胞异质性及发育进程中的基因调控奥秘提供了关键手段。
☞ ATAC-seq:染色质开放性与基因调控元件研究的先锋
ATAC-seq聚焦染色质开放性研究,可高效探索全基因组染色质开放区域与潜在调控元件,凭借样本需求量少、检测灵敏快速的优势,成为解析基因表达调控的重要技术。
☞ 技术组合:发挥协同优势
通常单一技术难以满足复杂研究需求,技术组合可提供更全面的研究视角。ATAC-seq与ChIP-seq、CUT&Tag或DAP-seq结合,能同时获取染色质开放性与蛋白质-DNA结合位点信息,帮助研究者深入解析基因调控网络。ATAC-seq与RNA-seq联合使用,可关联染色质开放性与基因表达变化,助力探索基因表达调控机制。
四、总结
DAP-seq、ChIP-seq、CUT&Tag、ATAC-seq四种技术在表观遗传研究中各展所长,其原理、特点与适用场景各异,我们需要结合具体研究需求和条件,才能做出合适的选择。欢迎感兴趣的同学前来咨询!
相关文章
更多 >

