长读长测序技术在过敏性疾病研究中的作用
2024-08-13 来源:本站 点击次数:1438
什么是过敏性疾病?
过敏性疾病,也被称为变态反应性疾病,是由人体免疫系统对某些外界物质产生异常反应而引起的一类疾病。当人接触到过敏原时,会在血液中产生一种特异性免疫球蛋白E抗体(IgE),对某种特定的过敏原产生过敏反应。过敏性疾病的发生具有遗传倾向,且近年来患病率呈上升趋势。
过敏性疾病根据病因,可分为以下几类:
1.呼吸道过敏性疾病:过敏性咽炎、过敏性鼻炎、哮喘等;
2.皮肤过敏性疾病:荨麻疹、湿疹、药疹、接触性皮炎等;
3.消化道过敏性疾病:食物蛋白诱导的小肠结肠炎综合征、食物蛋白诱导的肠病等;
4.眼部过敏性疾病:过敏性结膜炎;
5.全身系统性过敏性疾病:过敏性休克、过敏性紫癜等。
长读长测序技术:助力解锁过敏性疾病研究
纳米孔测序等长读长测序技术以其超长的读长、实时检测和直接检测碱基甲基化修饰等优势,在医学及生命科学领域发挥着重要作用。这项技术能够完整组装拼接物种全基因组,发现新的过敏源,并且能深入分析个体的基因组,识别与过敏性疾病相关的遗传变异,为疾病的预防、诊断和治疗提供科学依据。
1、尘螨Dermatophagoides farinae染色体级别基因组和转录组组装揭示两种新过敏原Der f 37和Der f 39

尘螨(HDMs),特别是Dermatophagoides pteronyssinus(Der p)、Dermatophagoides farinae(Der f),是吸入性过敏原的主要来源。需要深入分析HDM过敏原的全谱,以阐明HDM过敏机制并指导诊断和免疫治疗的发展。
准确的尘螨(HDM)基因组和转录组数据将促进我们对HDM过敏原的理解。本研究旨在通过进行尘螨(Dermatophagoides farinae)的染色体级基因组组装和精确的转录组分析,鉴定新的过敏原。在本研究中,从HDM虫体和卵中提取遗传物质,使用下一代测序(NGS)的短读长序列和PacBio/Nanopore测序的长读长序列来构建D. farinae的核基因组、转录组和线粒体基因组。通过与WHO/IUIS过敏原数据库中的氨基酸序列进行比对,筛选出候选同源物。
结果显示,与D. farinae草图基因组相比,本研究中获得的测序读段中来源于细菌DNA的占比大幅降低(从22.9888%降至1.5585%),基因组大小得到校正(从53.55 Mb增至58.77 Mb),并且组装获得的连续片段(contig)N50增加(从8.54 kb增至9365.49 kb)。组装的基因组包含10个contig,极低的微生物污染,鉴定得到33个典型过敏原和2个新的过敏原。克隆了8个同源性≥50%的同源物,其中2个与HDM过敏血清结合,被鉴定为过敏原(Der f 37和Der f 39)。综上,本研究获得了D. farinae的染色体级基因组、转录组和线粒体基因组,能够支持过敏原鉴定和诊断及免疫治疗疫苗的开发。
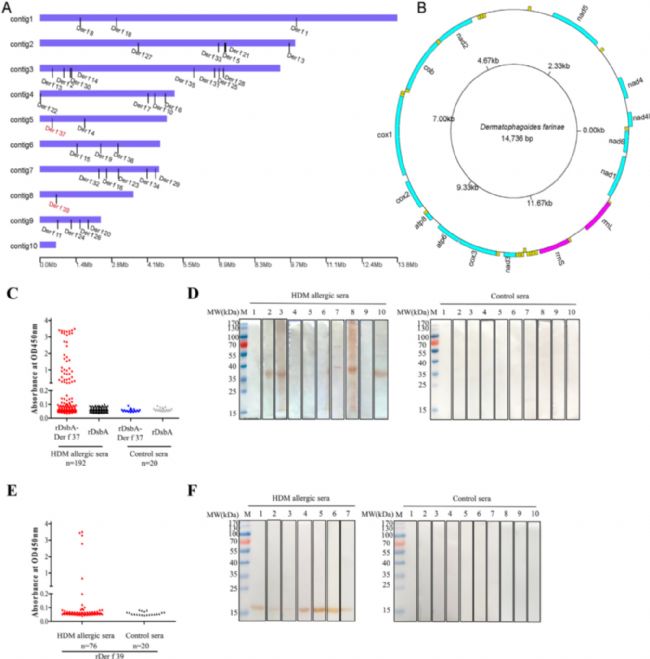
Fig.1 Chromsome-level assembly of genome and mitochondrion genome of D. farinae and identification of novel HDM allergens Der f 37 and Der f 39. A. Genome assembly showing locations of allergen genes. B. Mitochondrial genome of D. farinae. Mite allergens based on WHO/IUIS allergen nomenclature (http://www.allergen.org/search.php?TaxSource¼Animalia%20Arthropoda). Der f 17 sequence information: not available; HDM allergen Groups 12 and 19: not found; Der p 38 homolog in D. farinae (bacterial lytic enzyme like protein, GenBank accession No. MT360919.1) showed no IgE-binding activity in this study. C. IgE binding activity determined by IgE-western blots of rDsbA-Der f 37 with individual sera from 192 HDM-allergic patients and 20 healthy non-allergic individuals. D. Western blot assay identifying rDer f 37 protein binding by IgE in sera from 10 patients with HDM allergies (left) and 10 non-HDM allergic subjects (control, right). E. IgE binding activity determined by IgE-ELISA of rDer f 39 with individual sera from 76 HDM-allergic patients and 20 healthy nonallergic individuals. F. Western blot assay identifying rDer f 39 protein binding by IgE in sera from 7 patients with HDM allergies (left) and 10 non-allergic subjects (control, right). The HDM-specific IgEs (>100 kUA/L) within the sera samples were evaluated using an ImmunoCAP system.
使用Nanopore测序技术,除了Contig2外,所有contig都超过了2Mb,最长的一个超过了13Mb,表明组装质量达到了接近染色体级别。本研究注释了组装基因组中的33个典型过敏原基因,包括2个新发现的蛋白质,即Der f 37和Der f 39,并确定了它们在相应contig中的位置。为了获得高质量的基因集,我们对染色体级组装结果进行了同源性分析、下一代RNA测序(RNA-seq)和基因组从头注释。研究者鉴定了10,684个蛋白质编码基因,并通过NCBI非冗余蛋白、SwissProt和KEGG数据库对超过91.67%的基因进行了功能注释。
在这项研究中,纳米孔测序技术发挥了至关重要的作用,它通过提供长读长序列数据,帮助研究者实现了对尘螨Dermatophagoides farinae基因组的染色体级别组装。这种长读长测序技术显著减少了组装过程中的缺失,提高了基因组的连续性和准确性,从而使得基因组大小的校正、组装序列N50的显著提升以及基因组中过敏原的精确鉴定成为可能。此外,纳米孔测序技术还有助于减少细菌DNA的污染,这对于提高基因组数据的质量至关重要,进而支持了过敏原的识别和诊断及免疫治疗疫苗的开发。
2、美洲大蠊基因组组装和注释揭示全面的蟑螂过敏原谱

美洲大蠊是城市地区最常见的蟑螂类型之一,已被报道有增加过敏和哮喘的风险。由于缺乏基因组相关信息,这一物种中只鉴定出了有限的过敏原组(Per a 1–13)。
为了扩展美洲大蠊的过敏原谱,研究团队采用基因组、转录组和蛋白质组方法,基于蛋白质同源性鉴定了潜在过敏原,并通过酶联免疫吸附试验(ELISA)在对美洲大蠊过敏的泰国患者中进行了测试。
研究结果表明,成功组装了染色体级别的美洲大蠊基因组(3.06 Gb),并显著提高了其连续性(N50 = 151 Mb)。鉴定了七组新的过敏原组,包括烯醇化酶(Per a 14)、细胞色素C(Per a 15)、丝束蛋白(Per a 16)、α-微管蛋白(Per a 17)、环肽(Per a 18)、孔蛋白3(Per a 19)和过氧化物酶6(Per a 20),并在Per a 7中鉴定了一个新的同种型变体(Per a 7.02)。此外,通过比较分析,揭示了与近缘的蜚蠊目物种之间可能存在的交叉反应性。
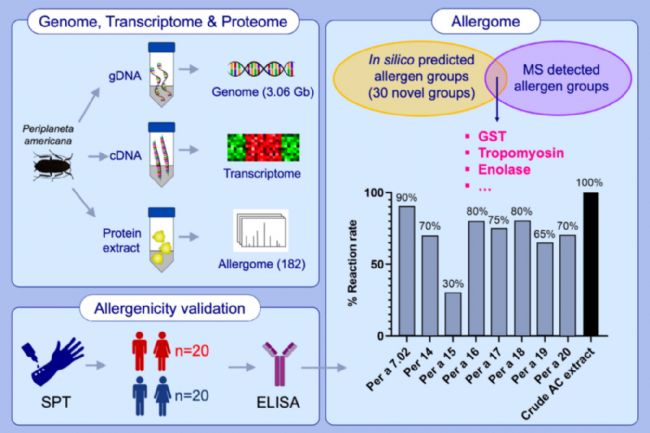
Our high-quality genome (3.06 Gb) and precisely annotated proteome (29,939) of the American cockroach help revealed a broader allergen spectrum. Seven novel allergen groups Per a 14 to Per a 20 and one isoallergens (Per a 7.02) were validated using ELISA in Thai cohort.
Comparative analysis of P. americana with the closely related Blattodea species revealed the possibility of cross-reaction.
Abbreviations: ELISA, enzyme-linked immunosorbent assay; gDNA, genomic DNA;
GST, glutathione S-transferases; SPT, skin prick test; MS, mass spectrometry
在这项研究中,纳米孔测序技术提供了长读长序列数据,有助于提升基因组组装的质量,尤其是在解决基因组中的重复区域和复杂结构方面。通过与Illumina高通量测序和Hi-C技术相结合,纳米孔测序技术使得研究者能够构建出一个更高连续性的基因组,这对于后续的过敏原发现和表征至关重要。这项研究的发现对于理解蟑螂引起的过敏疾病、开发新的诊断方法和特异性免疫疗法具有重要意义。
3、RNA测序在乳糜泻研究中的应用综述

近几十年来,RNA测序(RNA-seq)技术取得了显著进步,成为转录组分析的关键技术。从常规细胞群体测序(bulk sequencing)向单细胞和空间方法的转变,将研究的精确度提高到了细胞层面,为理解个体免疫细胞及自身免疫病发展的分子机制提供了宝贵信息。在乳糜泻(CeD)等自身免疫疾病研究中,RNA-seq技术尤为关键,广泛应用于转录组比较、营养基因组学、小麦研究、CeD背景下的癌症研究、基因与非编码RNA介导的表观遗传研究、疾病监测与生物标志物发现、线粒体功能调控、治疗靶点识别和药物作用机制等多个领域。
长读长cDNA测序和直接RNA-seq能提供更全面的亚型水平数据,尤其擅长识别短读长方法难以检测的亚型,在该研究领域正逐渐取代短读长测序。相对于使用传统NGS短读长测序面临的挑战,基于PacBio长读长测序技术为研究麸质蛋白基因提供了解决方案,能够精确获取mRNA完整序列信息,结合RNA测序、蛋白质组学分析和高级生物信息学工具,可更深入了解复杂麸质蛋白及面包小麦中的乳糜泻表位,促进麸质蛋白作用研究并支持更健康小麦品种的开发。
长读长测序技术在过敏性疾病研究的未来展望
长读长测序技术因其明显的优势特点,在过敏性疾病的研究中具有很大的应用潜力:
- 基因组变异检测:长读长测序技术能够检测到基因组中的结构变异,包括插入、缺失、倒位和复制等,这些变异可能与过敏性疾病的发生有关。
- 免疫组库分析:过敏性疾病与免疫系统的反应密切相关,长读长测序可以用于免疫组库的分析,帮助研究B细胞和T细胞受体的多样性,进而了解免疫应答的复杂性。
- 微生物组分析:肠道微生物组与过敏性疾病有关,长读长测序有助于解析微生物群落的组成和功能,为研究过敏性疾病的微生物组基础提供数据支持。
- 表观遗传学研究:DNA甲基化等表观遗传修饰可能影响过敏性疾病的发生发展,长读长测序技术能够直接检测这些修饰,有助于揭示表观遗传在过敏性疾病中的作用。
- 快速病原体检测:对于由病原体引起的过敏性疾病,长读长测序技术可以快速识别病原体的基因组序列,有助于病原体的鉴定和疫情监控。
- 药物基因组学研究:长读长测序技术有助于研究药物代谢酶和药物靶点的基因多态性,为过敏性疾病的个体化治疗提供信息。
- 疾病机理研究:长读长测序技术可以揭示过敏性疾病相关的复杂基因调控网络和信号传导途径,增进对疾病机理的理解。
- 个性化医疗:通过长读长测序技术,可以为过敏性疾病患者提供更精准的基因组信息,有助于开发个性化的预防、诊断和治疗方案。
参考文献
[1]Chen J, Cai Z, Fan D, Hu J, Hou Y, He Y, Zhang Z, Zhao Z, Gao P, Hu W, Sun J, Li J, Ji K. Chromosome-level assembly of Dermatophagoides farinae genome and transcriptome reveals two novel allergens Der f 37 and Der f 39. World Allergy Organ J. 2021 Sep 28;14(10):100590. doi: 10.1016/j.waojou.2021.100590. PMID: 34659625; PMCID: PMC8487952.
[2]Wang L, Xiong Q, Saelim N, Wang L, Nong W, Wan AT, Shi M, Liu X, Cao Q, Hui JHL, Sookrung N, Leung TF, Tungtrongchitr A, Tsui SKW. Genome assembly and annotation of Periplaneta americana reveal a comprehensive cockroach allergen profile. Allergy. 2023 Apr;78(4):1088-1103. doi: 10.1111/all.15531. Epub 2022 Oct 5. PMID: 36153808.
[3]Shoaran M, Sabaie H, Mostafavi M, Rezazadeh M. A comprehensive review of the applications of RNA sequencing in celiac disease research. Gene. 2024 Jun 11;927:148681. doi: 10.1016/j.gene.2024.148681. Epub ahead of print. PMID: 38871036.
相关文章
更多 >

