利用单细胞、空间和原位综合分析技术绘制肿瘤微环境高分辨率图谱
2025-03-06 来源:本站 点击次数:600
影响因子:14.7
主要技术:scRNA-seq、Visium、Xenium
导语
传统单细胞测序技术虽能解析细胞异质性,但缺乏空间信息;而空间技术分辨率有限,难以精确定位细胞间相互作用。本文中,研究人员利用单细胞、空间和原位技术,对FFPE保存的乳腺癌组织进行研究。运用scFFPE-seq、Visium CytAssist和Xenium In Situ技术,分析肿瘤异质性(图1)。通过整合这些技术,不仅能更准确地注释细胞类型,还发现了新的细胞类型和分子特征。在两个肿瘤样本中,分别识别出三受体阳性细胞和边界细胞,并获取其转录组信息。这一研究为深入理解肿瘤微环境的细胞网络提供了新视角,展示了多模态技术整合在肿瘤研究中的巨大潜力。
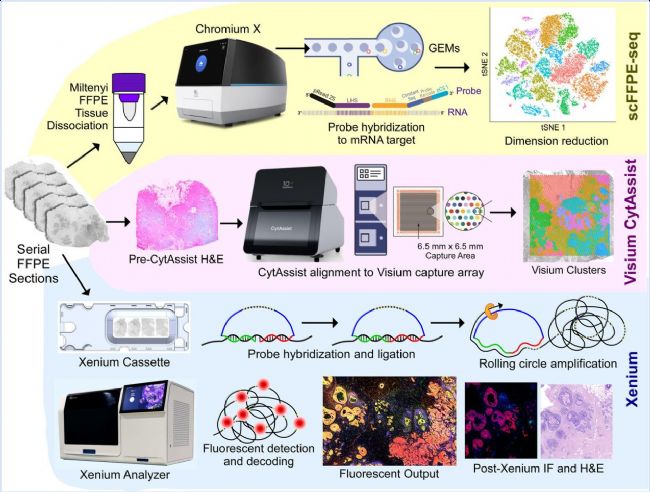
图 1
主要技术
scRNA-seq、Visium、Xenium
研究结果
1. 人乳腺癌FFPE组织进行综合绘图
对乳腺癌FFPE样本进行单细胞转录组测序(scFFPE-seq),从乳腺癌组织中生成了单细胞转录组数据,分析获得17个群(图2a)。通过收集与用于scFFPE-seq的组织切片相邻的5μm组织切片来生成Visium转录组数据,同样获得了17个群(图2b)。通过marker基因对scFFPE-seq数据进行注释,并将细胞类型映射到Visium数据上,其中十群注释成具体细胞类型或疾病状态,而其他七个群则由混合的细胞类型组成(图2b)。通过Visium确定了三个肿瘤结构域的空间位置,根据scFFPE-seq,这些肿瘤结构域显示为不同的集群,包括两种分子上不同类型的导管原位癌DCIS#1、DCIS#2和浸润性肿瘤。Visium结果还得出免疫细胞和间质细胞的大致区域(图2c)。
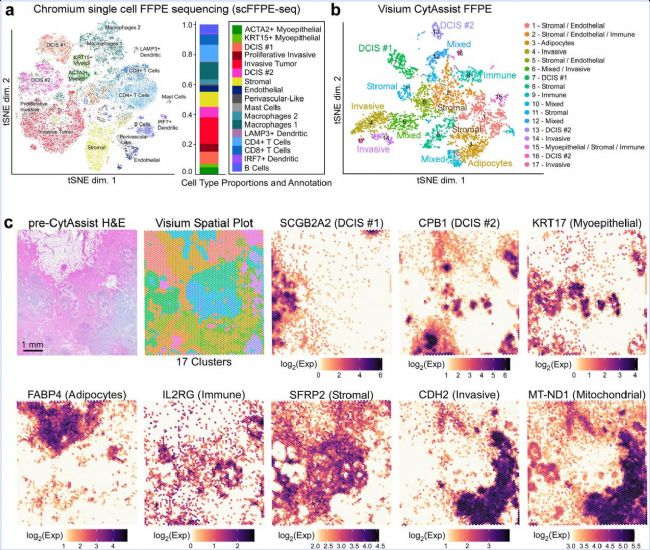
图 2
2. Xenium原位数据在单细胞水平上揭示肿瘤异质性
scFFPE-seq和Visium数据共同揭示了两种不同的DCIS和肌上皮细胞类型,研究人员运用Xenium技术以更详细的细节和更高的分辨率探索这些区域的临近细胞。Xenium 包含人类乳腺基因组(280个基因)和33个附加基因,共313个基因。对解码一个周期后观察RNA荧光图像,以高分辨率显示了组织的详细结构(图3a)。对组织的进一步分析能够从penal中选择相关基因,以鉴定基质细胞、淋巴细胞、巨噬细胞、肌上皮细胞等(图3b)。还进行 H&E染色(图3c),以便能够与病理学家的注释交叉参考。通过软件进一步可视化细胞分割边界(图3d),在分析的组织切片中,研究人员观察到共167,885个细胞和36,944,521个转录本(Q值≥20),每个细胞的中位转录本数量为166(图3e-f)。将scFFPE-seq数据下采样至313个基因后,发现scFFPE-seq每个细胞的中位基因数为34,而Xenium数据为62(图3g-h)。为验证Xenium人类乳腺癌面板的准确性,研究人员将scFFPE-seq注释转移到Xenium数据中,发现86%的细胞在Xenium数据中被明确识别为单一细胞类型(图3i)。这一结果证实了Xenium能够捕获生物学异质性。研究人员展示了基于Xenium数据的细胞类型注释结果,通过精确的转录本分配,识别出与单细胞数据相似的细胞类型,但是DCIS 亚型和增殖性肿瘤细胞没有得到解决(图3j-k)。这进一步证明了Xenium技术在单细胞水平上的可靠性和准确性。最后,研究人员生成了Xenium空间图,直观地展示了不同细胞类型在肿瘤微环境中的空间分布情况(图3l)。
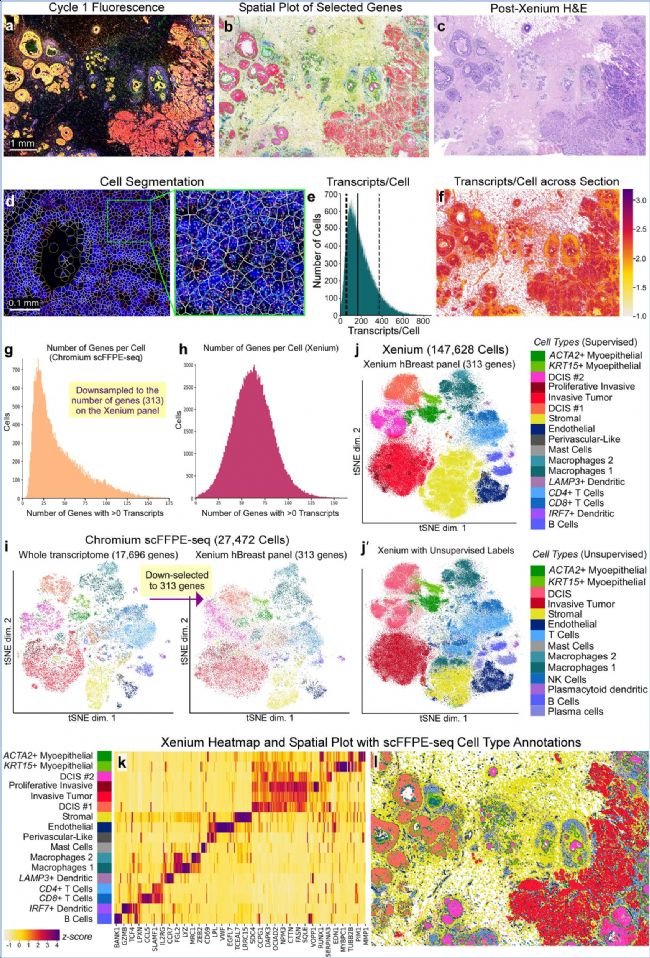
图 3
3. 具有三种肿瘤亚型的乳腺癌样本中细胞群体存在异质性
研究人员首先利用scFFPE-seq数据将三种不同的肿瘤上皮细胞亚型和两种肌上皮亚型映射到Xenium数据中,选择了三个感兴趣的区域(ROIs):DCIS #1、DCIS #2和浸润性肿瘤区域进行深入研究(图4a)。使用scFFPE-seq数据确定了15种细胞类型在ROIs中的比例,包括淋巴细胞、肌上皮细胞和浸润性细胞等。识别出四个主要的细胞类型组成差异:ACTA2+肌上皮细胞在DCIS #2 ROI中显著存在,在DCIS #1 ROI中较少,在浸润性ROI中缺失,浸润性肿瘤细胞在DCIS #2 ROI中存在,内皮细胞在浸润性ROI中数量稍多(图4b)。也进一步验证了这些细胞类型组成差异,并展示了DCIS #2 ROI中ACTA2+肌上皮细胞的边界环绕着浸润性细胞。这表明DCIS #2 ROI比DCIS #1 ROI更具侵袭性。浸润性ROI中浸润性细胞类型的发生率高,肌上皮细胞类型完全缺失。Xenium的高分辨率使得能够捕获邻近细胞的信息(图4c)。七种主要细胞类型的典型标记物和肿瘤亚型之间差异表达基因的表达分析揭示了MZB1是DCIS #1 ROI的特异性标记物,GJB2+基质细胞在DCIS #2 ROI中存在,ALDH1A3、KRT15和KRT23:在DCIS #1 ROI的肌上皮细胞中高表达,MMP12为巨噬细胞标记物,在浸润性ROI中缺失(图4d)。
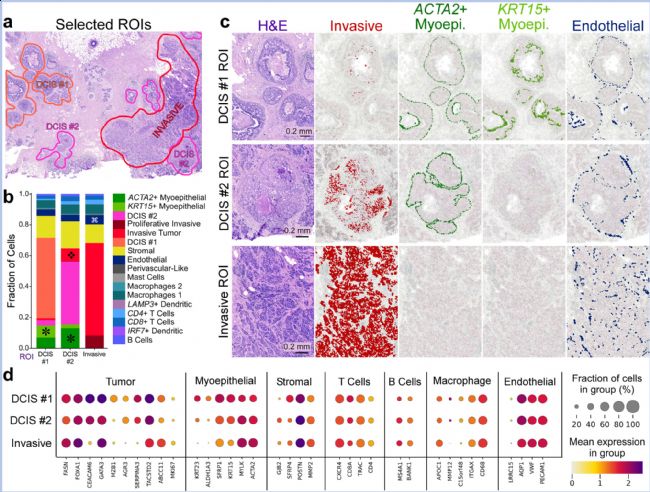
图 4
4. 整合空间转录组数据和靶向原位数据描述了三阳性区域
本研究使用的组织块被标注为HER2+/ER+/PR−。Xenium数据显示,大部分区域为ERBB2+(HER2+)和双阳性ERBB2+/ESR1+(HER2+/ER+)基因表达(图5a)。然而,研究人员发现了一个小的三阳性ERBB2+/ESR1+/PGR+(HER2+/ER+/PR+)DCIS ROI,该区域位于脂肪细胞附近,主要由DCIS #2肿瘤上皮细胞组成,缺乏KRT15+肌上皮细胞层(图5b-d)。接下来,研究人员比较了Xenium和scFFPE-seq数据中三种临床上相关的受体基因的表达。发现在scFFPE-seq数据中,PGR+细胞很少,且这些细胞不共表达ESR1或ERBB2(图5e-f)。在Visium数据中,三阳性区域仅由5-6个点表示,作为Cluster 12的一部分。发现Visium数据能够提供全转录组信息,揭示了三阳性区域的基因表达特征(图5g-h)。通过Xenium和Visium的数据,研究人员将Xenium中的转录本按邻近关系分配到Visium点上,并对这五个点与其他恶性点进行全转录组差异表达分析。识别出48个与PGR−DCIS #1相比的差异表达基因和44个与PGR−DCIS #2相比的差异表达基因(图5i-j)。
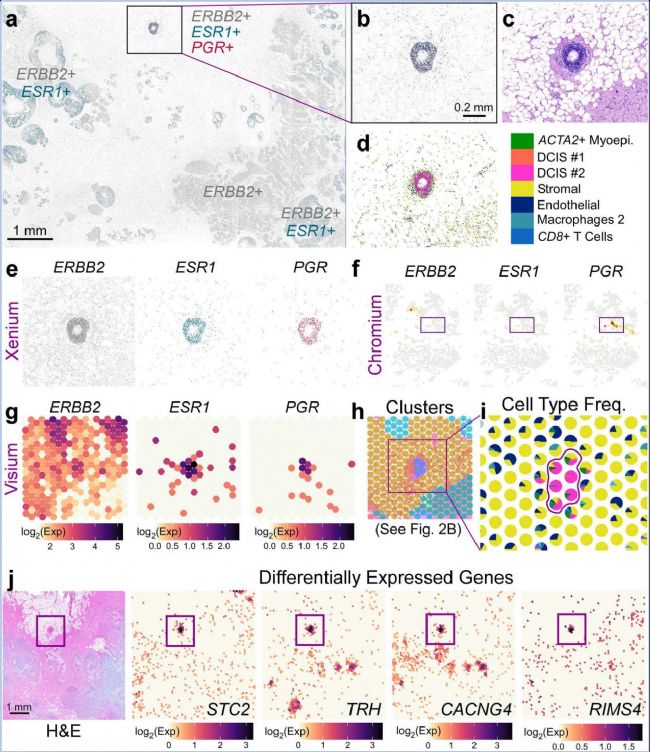
图 5
5. 整合单细胞和原位数据分析肌上皮边界的细胞
为探索数据整合策略是否可以应用于不同生物样本中DCIS向浸润性过渡的研究。研究人员用不同的人乳腺癌切片进行验证,该样本被标注为HER2+/ER−/PR−,包含正常、DCIS和浸润性区域。使用相同Xenium penal对样本相同处理,识别了免疫细胞、肌上皮细胞、上皮细胞和肿瘤细胞群体。在样本中检测到脂肪细胞、细胞毒性T细胞(CTLA4+)、浆细胞、树突状细胞等。两个基质细胞群体分离成不同的簇,并且在空间上不同。对肿瘤、肌上皮细胞和巨噬细胞群体进行亚群分析,识别了增殖性(TOP2A+)、SCD+和S100A8+肿瘤细胞,以及M1、M2和CD83+巨噬细胞。发现三种上皮细胞(ESR1+、PIGR+和OPRPN+)和一种肌上皮细胞(DST+)在分子和空间上都是不同的,与scRNA-seq参考数据一致。研究人员注意到一个位于肿瘤和肌上皮细胞群体之间的亚群,这些细胞共表达肿瘤(ERBB2、ABCC11)和肌上皮细胞(MYLK、DST)标记物(图6a)。并通过对DCIS区域的高倍镜检查,确认这些细胞确实共表达这两种标记物(图6b-c)。作为对照,研究人员观察了正常导管区域,其中肌上皮细胞和上皮细胞紧密相邻,但细胞类型特异性标记物没有混合,这表明观察结果不是由于粗略的分割错误(图6d)。接下来,研究人员对Xenium数据中识别的边界细胞群体进行了差异表达基因分析,并在样本1的scFFPE-seq数据中寻找这种细胞类型特异性的基因表达谱,发现这些细胞中的大多数之前被注释为肌上皮细胞,占scFFPE-seq数据中约1%的细胞。将这些细胞与肿瘤细胞和肌上皮细胞进行比较,并推导出全转录组信息。这导致识别出在边界细胞中高表达的基因CX3CL1、CCL28、PROM1等,这些基因在肿瘤细胞、肌上皮细胞或任何其他共定位细胞类型中不表达(图6e-f)。
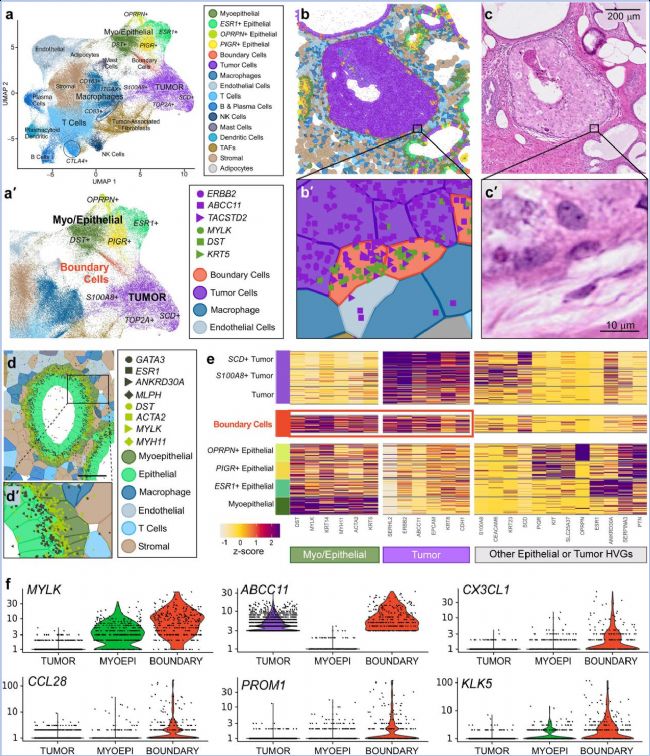
图 6
结语
本研究通过整合单细胞、空间和原位分析技术,对乳腺癌组织微环境进行了高分辨率的基因表达分析,揭示了肿瘤异质性和关键细胞类型的分子特征。研究使用了两个FFPE乳腺癌组织块,通过scFFPE-seq、Visium和Xenium技术,识别了不同的肿瘤亚型和罕见的边界细胞群体。在样本1中,研究人员发现了三阳性(ERBB2+/ESR1+/PGR+)DCIS区域,并识别了与侵袭性相关的分子差异。在样本2中,研究人员识别了一种罕见的边界细胞群体,这些细胞共表达肿瘤和肌上皮细胞标记物,可能在DCIS到浸润性癌的过渡中起关键作用。这些发现展示了多技术整合在揭示肿瘤异质性方面的强大能力,为理解乳腺癌的分子机制提供了新的见解,有助于推进肿瘤学研究和诊断治疗的发展。
参考文献:
Janesick A, Shelansky R, Gottscho AD, et al. High resolution mapping of the tumor microenvironment using integrated single-cell, spatial and in situ analysis. Nat Commun. 2023 Dec 19;14(1):8353. doi: 10.1038/s41467-023-43458-x. PMID: 38114474; PMCID: PMC10730913.

