在单细胞RNA测序中DoubletFinder双细胞分析方法详解
2025-05-14 来源:本站 点击次数:1869在单细胞RNA测序(scRNA-seq)中,当两个细胞被捕获到同一反应液滴时,会形成双联体(doublet)。在后续分析中会因带有相同的Barcode,会被认为是一个细胞的伪细胞。这类细胞会对分析产生影响,从而扭曲分析结果。因此,计算doublet检测方法成为必要工具。
《Benchmarking Computational Doublet-Detection Methods for Single-Cell RNA Sequencing Data》文献系统性评估了九种主流方法(包括doubletCells、Scrublet、cxds、bcds、hybrid、Solo、DoubletDetection、DoubletFinder和DoubletDecon),使用16个真实数据集,并通过scDesign生成的112个合成数据集,从检测的准确性,下游分析的影响,到计算效率等各方面评估。
综合显示,DoubletFinder在检测准确性上表现最佳。
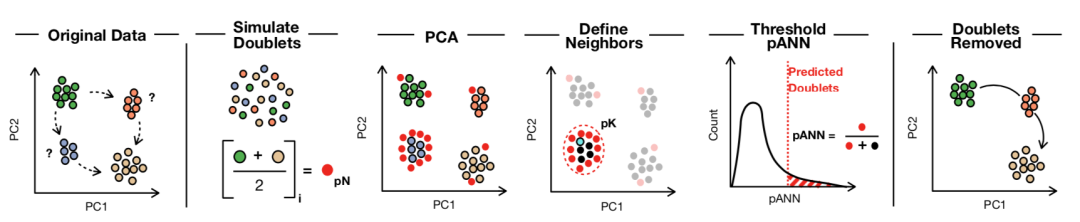
DoubletFinder分析可分为4个步骤:
(1) 利用已有的scRNA-seq数据生成doublet;
(2) 对合并的真实人工数据进行预处理;
(3) 进行PCA主成分分析,利用PC距离矩阵求出每个细胞的人工k个最近邻(pANN)的比例;
(4) 根据预期的doublet数量排序和计算阈值pANN值。
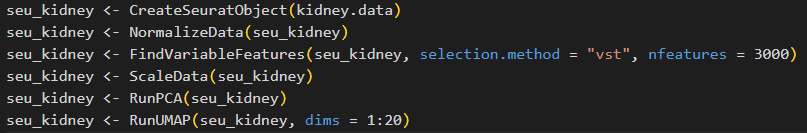
以下是我们分析的示例代码:
1.读入单细胞数据,并进行一系列预处理(包括单细胞数据质控、标准化、降维聚类等):
seu_kidney <- CreateSeuratObject(kidney.data)
seu_kidney <- NormalizeData(seu_kidney)
seu_kidney <- FindVariableFeatures(seu_kidney, selection.method = "vst", nfeatures = 3000)
seu_kidney <- ScaleData(seu_kidney)
seu_kidney <- RunPCA(seu_kidney)
seu_kidney <- RunUMAP(seu_kidney, dims = 1:20)
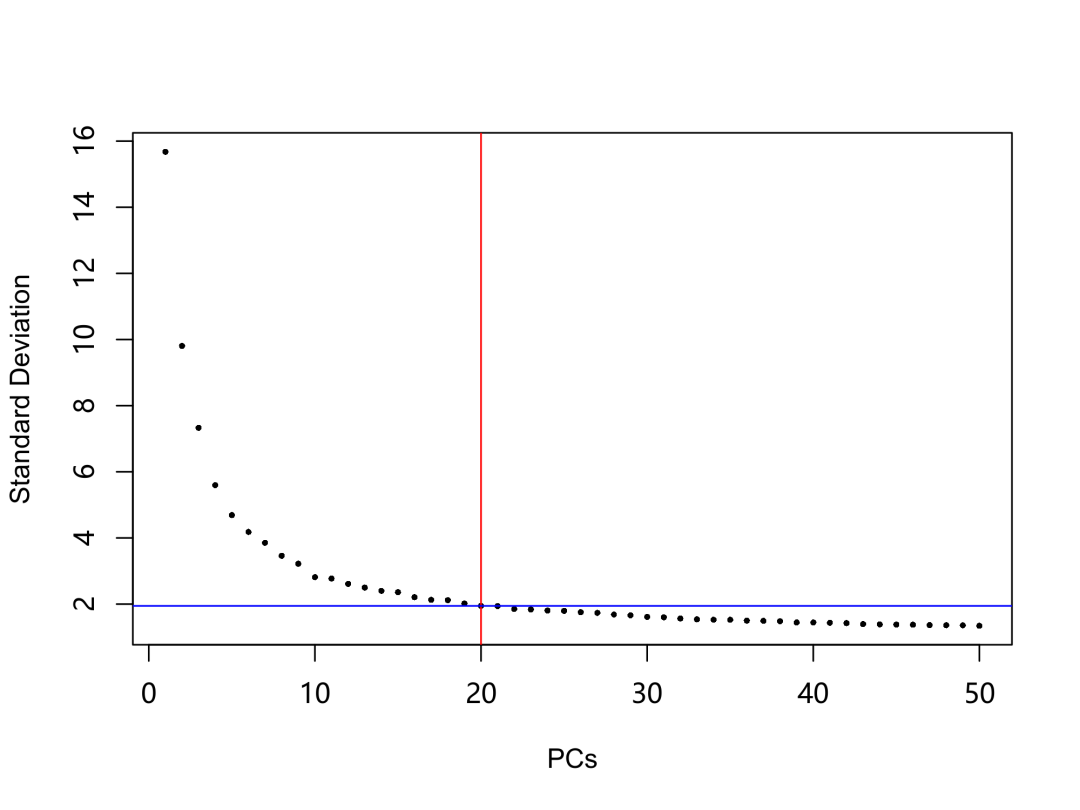
选择统计上显著的主成分的数量,下图为我们选择的主成分数量。
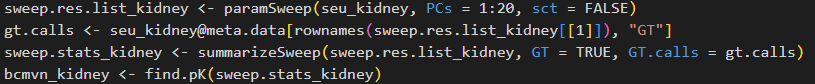
2. 寻找最优pK值:
sweep.res.list_kidney <- paramSweep(seu_kidney, PCs = 1:20, sct = FALSE)
gt.calls <- seu_kidney@meta.data[rownames (sweep.res.list_kidney[[1]]), "GT"]
sweep.stats_kidney <- summarizeSweep(sweep.res.list_kidney, GT = TRUE, GT.calls = gt.calls)
bcmvn_kidney <- find.pK(sweep.stats_kidney)
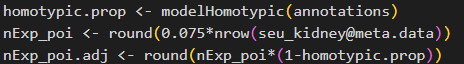
3. 双细胞比例计算:
homotypic.prop <- modelHomotypic(annotations)
nExp_poi <- round(0.075*nrow(seu_kidney@meta.data))
nExp_poi.adj <- round(nExp_poi*(1-homotypic.prop))
4. 鉴定双细胞:
seu_kidney <- doubletFinder(seu_kidney, PCs = 1:20, pN = 0.25, pK = 0.09, nExp = nExp_poi, reuse.pANN = FALSE, sct = FALSE)
seu_kidney <- doubletFinder(seu_kidney, PCs = 1:20, pN = 0.25, pK = 0.09, nExp = nExp_poi.adj, reuse.pANN = "pANN_0.25_0.09_913", sct = FALSE)

5. 确定了Doublet-Low Confidience, Doublet-High Confidience,Singlet三种细胞类型,并进行可视化,包含TSNE和UMAP两种展示形式:
DimPlot(pbmc, reduction = "tsne", group.by ="DF_hi.lo",cols =c("red","gold","#1bb3b6"),pt.size = 0.8) + ggtitle("DoubletFinder")
DimPlot(pbmc, reduction = "umap", group.by ="DF_hi.lo",cols =c("red","gold","#1bb3b6"),pt.size = 0.8) + ggtitle("DoubletFinder")

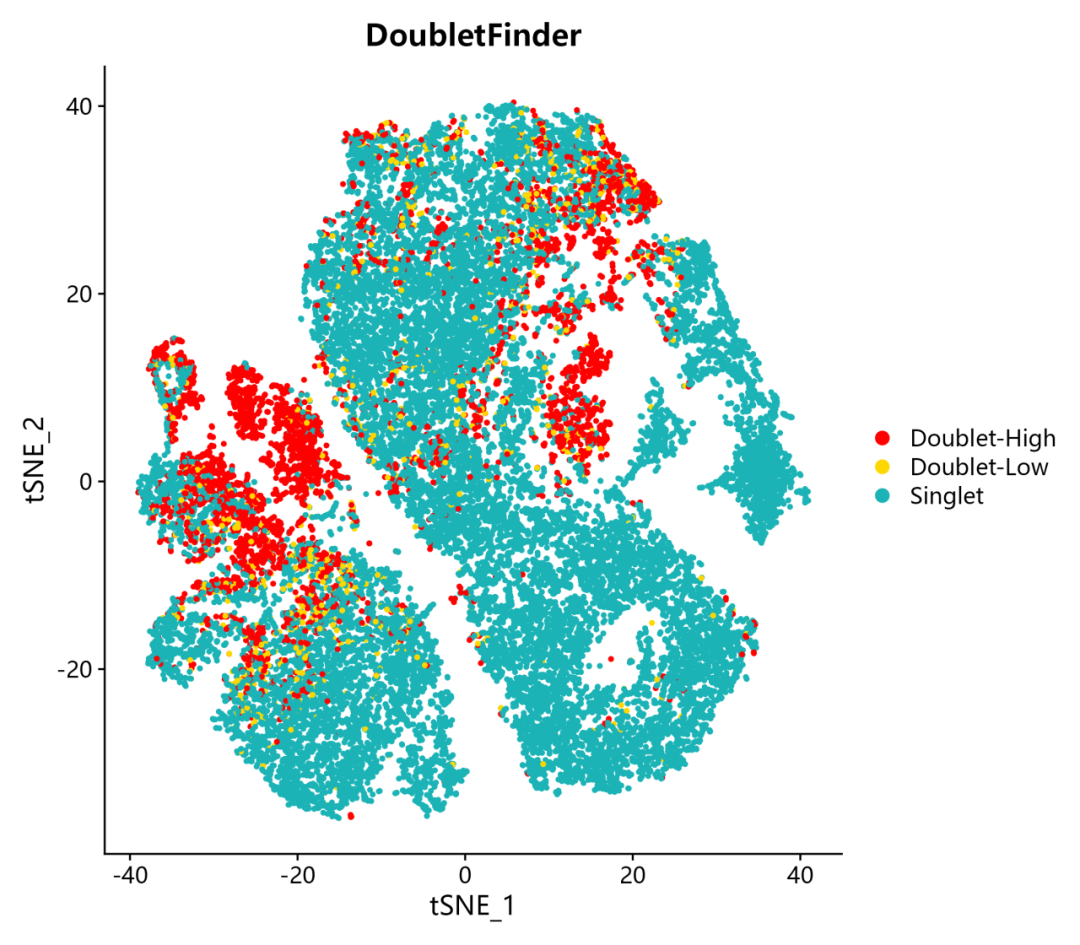
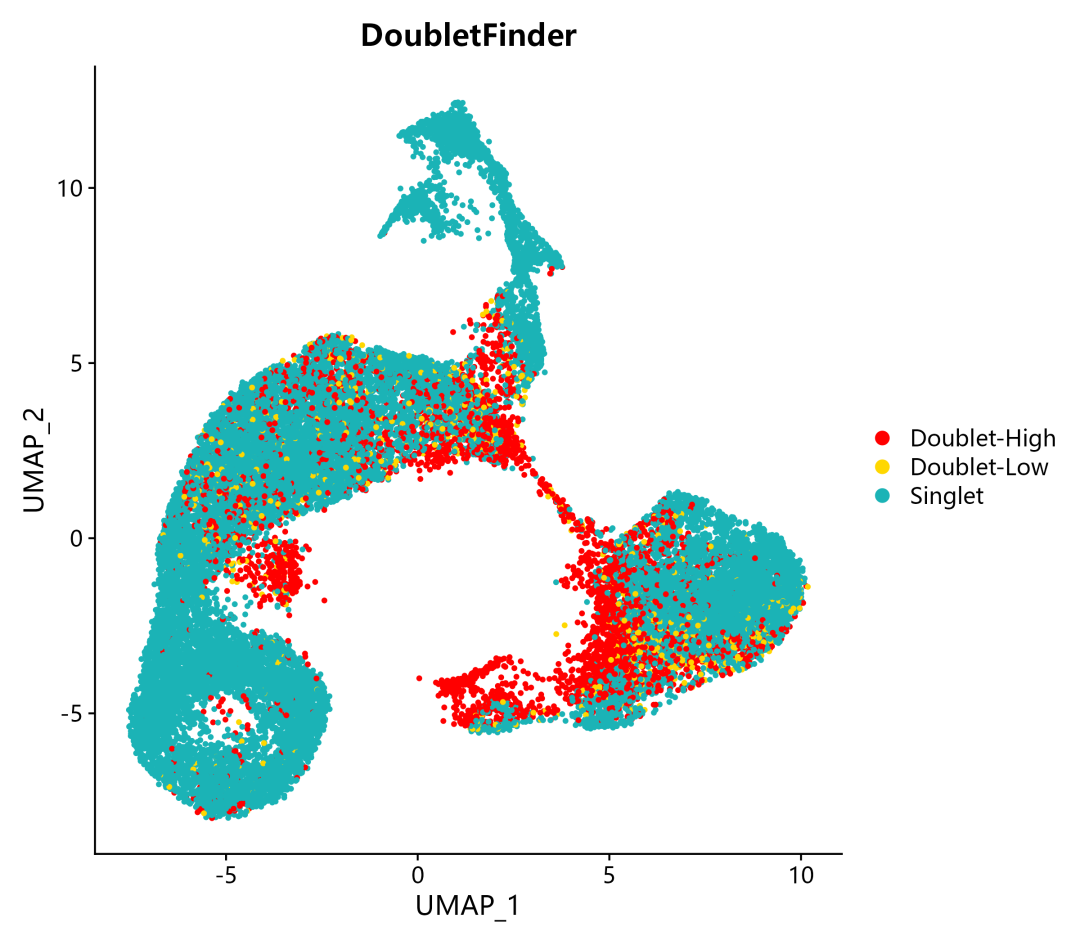
在完成DoubletFinder双细胞分析之后,我们可以从单细胞数据的meta.data表中,查看结果,用于之后的下游分析。
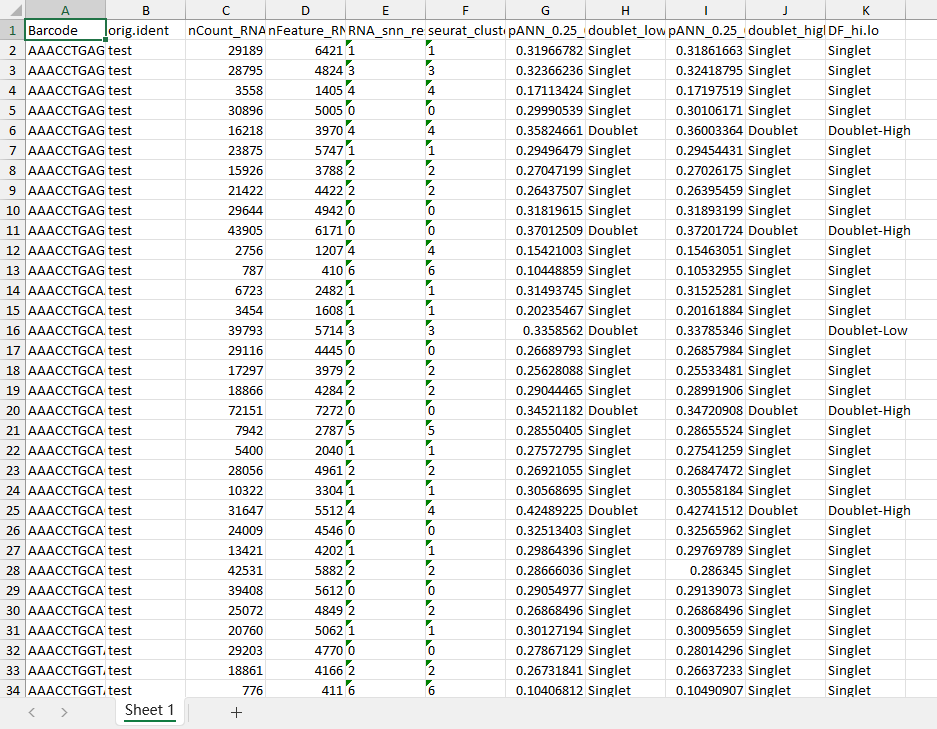
以上是对DoubletFinder双细胞分析方法的展示。双细胞的去除非常必要,当我们剔除这类潜在的双细胞数据后,后续的分析结果也就更可靠了。

