生物公共数据库使用中的常见问题和解决方案
2025-06-12 来源:本站 点击次数:990在生物学研究的"问题导向型"与"数据驱动型"两大主流范式中,前人已发表的组学数据正由"辅助材料"向"核心资源"转型。这些数据在验证假设、发现新规律、突破实验瓶颈等方面具有不可替代的作用:
1. 验证与纠偏
通过分析他人数据,可验证研究结果的普遍性和可重复性。
2.数据整合与统计效力提升
对于受限于样本量的研究(如罕见病研究),整合多源数据可显著增强研究结论的可信度。
3.科学新发现的"孵化器"
已发表数据中常蕴含未被关注的关联性。例如:从癌症基因组数据中发掘新突变热点,或通过跨物种比较揭示进化规律。
4.资源与伦理限制下的替代方案
针对高成本实验(如长期生态监测)或伦理敏感研究(如人类疾病模型),合理利用公开数据可显著缩短研究周期并降低成本。
尽管已发表数据价值显著,研究者在实际应用中常面临以下挑战:
01 文献里的数据找不到来源
文献通常在方法或结论部分设置"Data availability"板块,说明数据存储的数据库及对应ID(图1)。部分文献可能将相关信息置于附录或"STAR METHODS"中的"KEY RESOURCES TABLE"(图2)。对于作者使用的第三方数据,需通过数据引用文献追溯来源。
快速定位技巧:搜索“data availability”、“availability”、“access”、“accessible”、“raw data”、“processed”等关键词。若出现“Any additional data are available from the corresponding author upon reasonable request”等表述,则表明数据未公开。
 图1. 文献名称:Global characterization of T cells in non-small-cell lung cancer by single-cell sequencing
图1. 文献名称:Global characterization of T cells in non-small-cell lung cancer by single-cell sequencing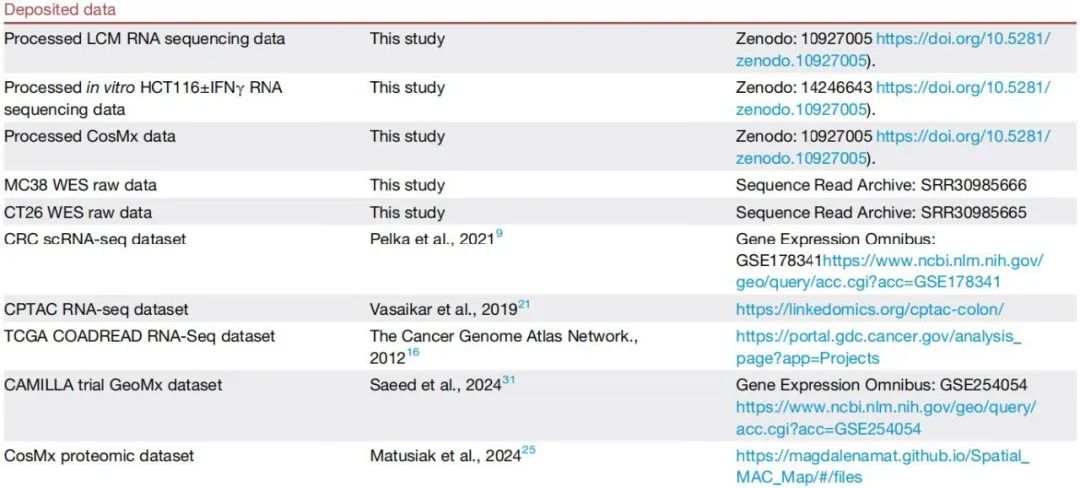 图2. 文献名称:A constitutive interferon-high immunophenotype defines response to immunotherapy in colorectal cancer
图2. 文献名称:A constitutive interferon-high immunophenotype defines response to immunotherapy in colorectal cancer
02 数据库界面复杂或全是英文,不知道该怎么操作
多数数据库因收录数据多样且功能复杂,导致界面复杂,加之全英文操作界面,显著增加学习成本。这也是我们撰写本系列文章的初衷,后续将逐一详解各类数据库的使用方法。
03 不知道该如何选择合适的数据库
不同的数据库里能下载到的数据也有各自特色。比如GEO数据库主要提供processed data(原始定量或标准化定量结果等),SRA专注原始测序数据。Zenodo数据库可能会包含中间数据(如R语言的rds格式)以及配套代码等。各位老师可以根据需求选择合适数据库。
04 数据库中的数据不知道该如何使用,使用时有什么注意事项
作者上传到数据库中的组学数据通常包含原始数据、原始定量(count)结果以及标准化后的定量结果等等。整合分析时有以下要点要注意:
1)参考基因组是否相同。敲黑板,画重点,合并分析前必须确认双方使用相同参考基因组版本。
2)选择遗传背景一致性较高的样本数据,可显著增强分析结论的重现性。
3)选择数据集时要注意定量分析软件和试剂的版本。比如10X Genomics公司单细胞转录组测序专用的定量分析软件Cell Ranger,早期版本的软件细胞鉴定策略和现版本有很大差别,实验试剂也在不断升级。如果下载使用的processed data是早期版本的结果,可能会因版本迭代造成差异,降低分析结果可靠性。
4)数据整合时要用相同的数据标准化方法。建议下载数据时使用原始定量而不是已经标准化后的定量结果。这里分享一个窍门。对于转录组测序数据,原始定量矩阵中基因表达值都是整数,而标准化后的结果一般是小数,可以根据这一点判断下载的数据类型。
5)有时我们下载到的定量结果基因名是数据库ID(比如人PTRPC基因在Ensembl数据库中的ID:ENSG00000081237)。数据库在更新时可能更改基因的ID,必要时进行ID转换。
6)当数据整合时发现PCA和样品相关性结果存在批次差异时,需要校正批次差。
用好公共数据,借助前人的成果加速你的研究。下期详解GEO数据库使用技巧,敬请期待!

