从多抗到单抗:精准测序技术解析多克隆抗体的转化与应用
2025-11-04 来源:本站 点击次数:606什么是多克隆抗体?
多克隆抗体(polyclonal antibodies, pAbs)是由异质性B细胞群体产生的抗体混合物,能够识别抗原表面的多个不同表位,因而具备多特异性特征[1]。相比之下,单克隆抗体(monoclonal antibodies, mAbs)来源于单一B细胞克隆,仅针对抗原的某一特定表位,展现出高度单一特异性[2]。
多克隆抗体相对于单克隆抗体的优势有哪些?
多克隆抗体的优势在于其能够同时识别抗原的多个表位,在抗原表位信息不完整或抗原存在高度异质性的研究场景中,表现出更优的抗原捕获能力和检测灵敏度。这一特性使其在临床初步筛查、早期诊断及基础研究领域具有不可替代的价值[3]。此外,多克隆抗体兼具生产成本优势和灵敏检测特性,使其在免疫印迹(Western Blot)和免疫沉淀(Immunoprecipitation)等关键技术中得到广泛应用。
多克隆抗体目前遇到的挑战有哪些?
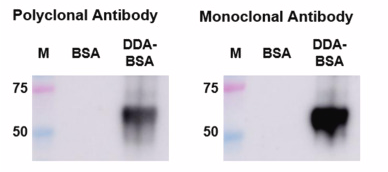
多克隆抗体制备过程中的核心挑战在于批次间差异性,其根源在于免疫动物的个体生物学差异,包括遗传背景、免疫状态及健康状况等因素。尽管通过标准化生产流程可在一定程度上降低工艺带来的相关变异,但无法完全消除生物体固有差异所导致的质量波动。因此,从多克隆抗体库中筛选获得高特异性、高灵敏度的单克隆抗体具有重要的产业转化价值。如图所示,在Western blot分析中,多克隆抗体与DDA-BSA结合物反应呈现多个条带,证实其多表位识别特性;而单克隆抗体仅显示单一显著条带,体现了其高度特异性[4]。
综上所述,尽管多克隆抗体在成本效益和多表位识别方面具有独特优势,但其批次间差异性仍是限制其应用的显著瓶颈。为提升抗体检测的准确性与结果可重复性,从多克隆抗体中定向筛选和开发高特异性单克隆抗体已成为行业共识,这一趋势也显著推动了抗体工程技术创新与临床转化应用的快速发展[5]。
什么是多克隆抗体测序?
BSI / 百蓁生物多克隆抗体测序是一种基于质谱从头测序、AI数据解析等多技术发现平台,能够从复杂抗体样本中精准获取单克隆抗体的完整序列信息的技术。该技术依托AI质谱驱动,可高效鉴定高特异性、高活性的单克隆序列,从而彻底解决多克隆抗体批次间的一致性问题,显著提升实验的准确性和可重复性。
多克隆抗体测序的应用场景有哪些?
1.生物药研发
2.体外诊断试剂开发
3.多抗转化成单抗或者单抗混合物
多克隆抗体测序有哪些优势?
01 更直接:
直接从样本中精准获取单克隆抗体完整序列
02 更精准:
AI质谱结合蛋白从头测序等多种先进技术,确保序列的100%准确,提升实验的高重复性。
03 更节省:
可实现无动物体外生产,同时明显提高产品的批间一致性和生产规模,更节省成本支出。
04 更快速:
最快一个月拿到单克隆抗体序列信息,通过体外的大规模表达纯化,大大缩短研发和生产周期
BSI / 百蓁生物多克隆抗体测序技术流程
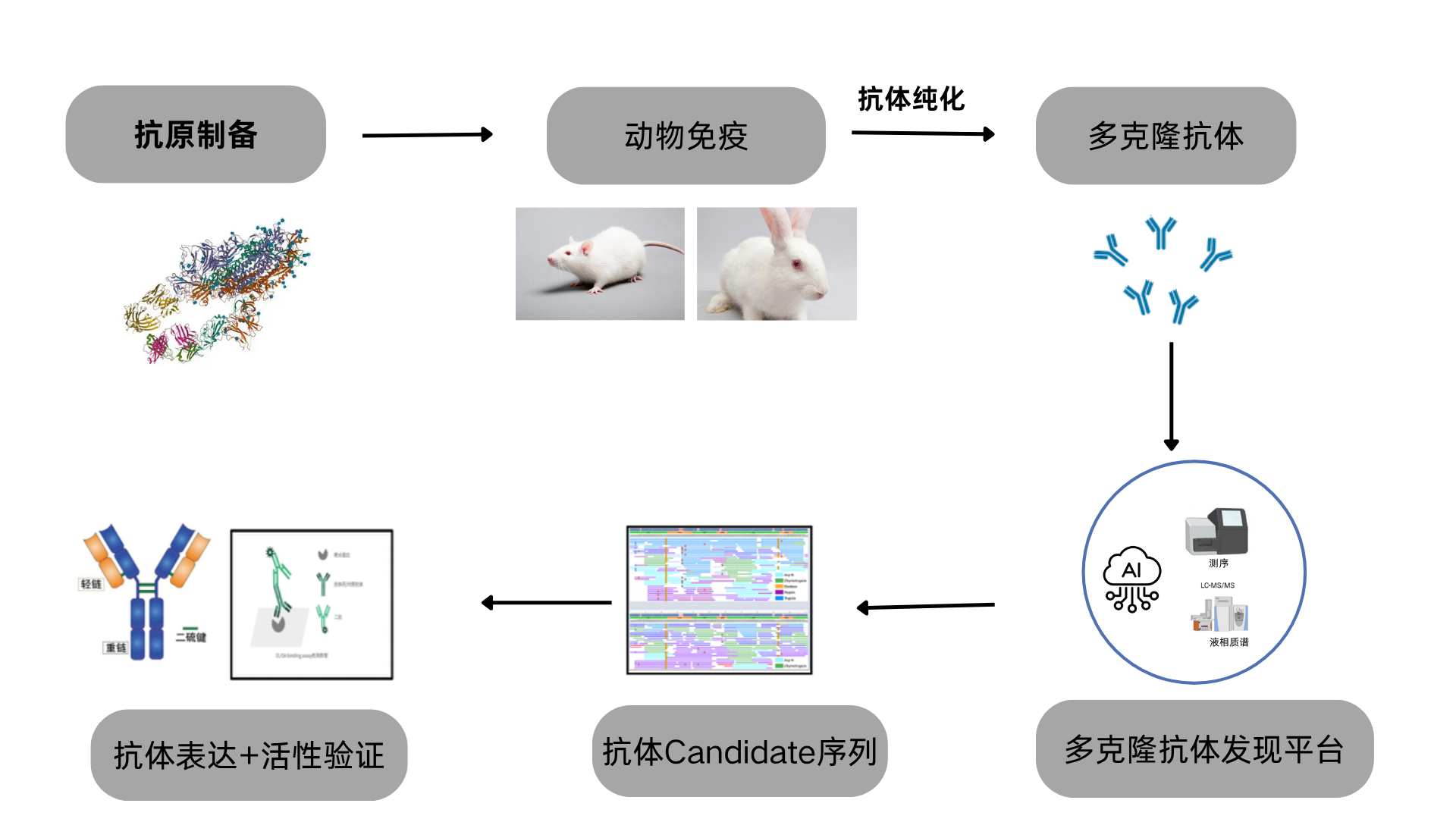
BSI / 百蓁生物提供基于AI质谱的一站式多克隆抗体测序发现服务
送样要求:
●新鲜脾脏组织1只(鼠或兔)或 1-5mL全血 或 2e6-1e7 新鲜提取的PBMC细胞
●0.1mg以上抗原特异性多抗溶液(1ug/uL以上)
●1mg高纯度抗原
交付标准&服务周期:
●100%覆盖高可信的完整抗体可变区序列 (5个以上)(1-1.5个月)
●100%覆盖高可信有活性的完整抗体序列 (5个以上)(2-2.5个月)
●100%覆盖高可信有活性的100mg单克隆抗体(5个以上 )(2.5-3个月)
成功案例
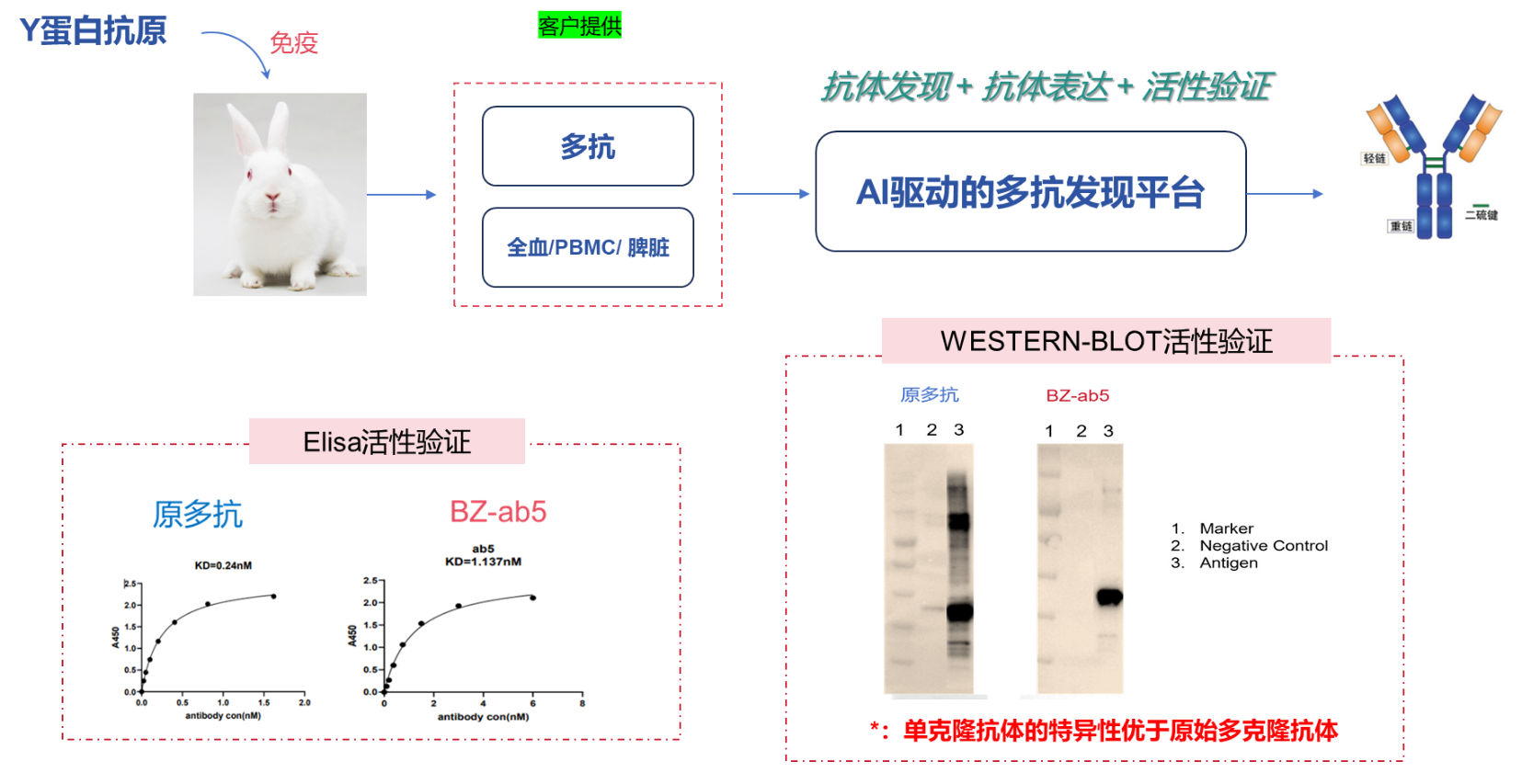
Y蛋白兔多克隆抗体中高特异性单克隆抗体发现
Y蛋白作为抗原免疫新西兰大白兔后,获得高特异性多克隆抗体,同时获取全血PBMC测序获得BCR序列模版。使用我司AI驱动多抗发现平台提供的抗体发现和活性验证全流程服务后,获得3个高活性的单克隆抗体。ELISA实验验证其中ab5具有与原多抗相似的亲和力,WESTERN-BLOT实验确证该单克隆抗体特异性明显优于原始多克隆抗体。
参考文献
[1] Tran N H , Zhang X , Xin L ,et al.De novo peptide sequencing by deep learning[J].Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(31):201705691.DOI:10.1073/pnas.1705691114.
[2] Zeping M , Ruixue Z , Ming L L .Mitigating the missing-fragmentation problem in de novo peptide sequencing with a two-stage graph-based deep learning model[J].Nature Machine Intelligence, 2023, 5(11):1250-1260.DOI:10.1038/s42256-023-00738-x.
[3] Li, K., Shan, B., Xin, L. et al. Proteoform search from protein database with top-down mass spectra. Nat Comput Sci (2025). doi.org/10.1038/s43588-025-00880-z
[4] Xin L, Li W, Zhang S, Tran NH, Chen Z, Ma J, Peng C, Aihemaiti A, Hoffman K, Zhang X, Sun W, Li L, Wang Z, Li M, Shan B. Sequencing of Polyclonal Antibodies by Integrating Intact Mass, Middle-Down and De Novo Bottom-Up Mass Spectrometry. Mol Cell Proteomics. 2025 Oct 13:101088. doi: 10.1016/j.mcpro.2025.101088. Epub ahead of print. PMID: 41093274.

